CAPÍTULO 8 Desenvolvimento dos Linfócitos e Rearranjo dos Genes dos Receptores de Antígenos
Os linfócitos expressam receptores de antígenos altamente diversos, capazes de reconhecer uma ampla variedade de substâncias estranhas. Essa diversidade é gerada durante o desenvolvimento dos linfócitos B e T maduros de células precursoras que não expressam receptores de antígenos e, portanto, são incapazes de reconhecer antígenos e a responder a eles. O processo pelo qual os progenitores dos linfócitos no timo e na medula óssea se diferenciam em linfócitos maduros que residem nos tecidos linfoides periféricos é denominado desenvolvimento dos linfócitos ou maturação dos linfócitos. O repertório imune é constituído pelo conjunto de receptores de antígenos – e, portanto, especificidades – expressos pelos linfócitos B e T, que são produzidos durante a maturação dessas células. A maturação é iniciada por sinais de receptores de superfície celular, que desempenham dois papéis principais – promovem a proliferação dos progenitores e também induzem a expressão de fatores de transcrição que atuam em conjunto para iniciar o rearranjo dos genes dos receptores de antígenos específicos e comprometer as células em desenvolvimento em uma célula B ou T. O rearranjo dos genes dos receptores de antígenos constitui um evento essencial no comprometimento de uma célula progenitora para um destino linfoide.
Este capítulo começa descrevendo o processo de comprometimento com as linhagens de linfócitos B e T e são discutidos alguns princípios e mecanismos comuns de desenvolvimento dessas células. A seguir, faz-se uma descrição dos processos exclusivos da maturação das células B e, por fim, daqueles peculiares ao desenvolvimento das células da linhagem dos linfócitos T.
VISÃO GERAL DO DESENVOLVIMENTO DOS LINFÓCITOS
A maturação dos linfócitos B e T envolve uma série de eventos que ocorrem nos órgãos linfóides geradores (Fig. 8-1). Esses eventos incluem os seguintes:
• O comprometimento de células progenitoras com a linhagem de células B ou T.
• A proliferação de progenitores e células comprometidas imaturas em estágios iniciais específicos do desenvolvimento, proporcionando um grande reservatório de células capazes de gerar linfócitos úteis.
• O rearranjo sequencial e ordenado dos genes dos receptores de antígenos e a expressão das proteínas receptoras de antígenos.
• Eventos de seleção preservam as células que expressam receptores de antígenos corretos e eliminam células potencialmente perigosas cujos receptores reconhecem de modo intenso os antígenos próprios. Esses pontos de controle durante o desenvolvimento asseguram que os linfócitos que expressam receptores funcionais com especificidades úteis irão amadurecer e entrar no sistema imune periférico.
• A diferenciação de células B e T em subpopulações funcionais e fenotipicamente distintas. As células B diferenciam-se em células foliculares, da zona marginal e em células B-1, enquanto as células T diferenciam-se em linfócitos T CD4+ e CD8+ e em células T γδ. Essa diferenciação em classes distintas fornece a especialização que é uma importante característica do sistema imune adaptativo.
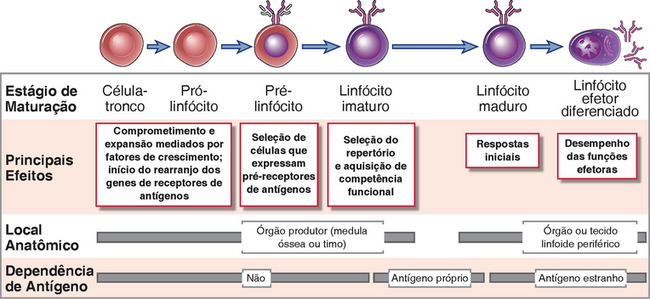
FIGURA 8-1 Estágios de maturação dos linfócitos. O desenvolvimento dos linfócitos B e T envolve a sequência dos estágios de maturação mostrada aqui. A figura ilustra a maturação das células B, porém os estágios básicos de maturação das células T são semelhantes.
A seguir, serão descritas as características de cada evento que é comum em ambas as linhagens de linfócitos B e T.
Comprometimento com as Linhagens de Células B e T e Proliferação dos Progenitores
As células-tronco pluripotentes na medula óssea (e no fígado fetal), denominadas células-tronco hematopoiéticas (CTH), dão origem a todas as linhagens de células sanguíneas, incluindo os linfócitos. As CTH amadurecem e transformam-se em progenitores linfoides comuns, que podem dar origem às células B, células T, células NK e algumas células dendríticas (Fig. 8-2). A maturação das células B de progenitores que se comprometem com essa linhagem ocorre, em sua maior parte, na medula óssea, e antes do nascimento, ocorre no fígado fetal. As células-tronco derivadas do fígado fetal dão origem principalmente a um tipo de célula B, denominada célula B-1 (mais bem definida em roedores), enquanto as CTH derivadas da medula óssea dão origem à maior parte das células B circulantes (células B foliculares). Os precursores linfócitos T deixam o fígado fetal antes do nascimento e a medula óssea durante a vida, circulando até o timo, onde completam o seu processo de maturação. A maioria das células T, que consistem em células T αβ, desenvolve-se de CTH derivadas da medula óssea, enquanto a maior parte das células T γδ origina-se de CTH do fígado fetal. Em geral, as células B e T geradas no início da vida fetal possuem um repertório de receptores de antígeno menos diverso. Apesar de suas localizações anatômicas diferentes, os eventos iniciais de maturação de ambos os linfócitos B e T são fundamentalmente semelhantes.
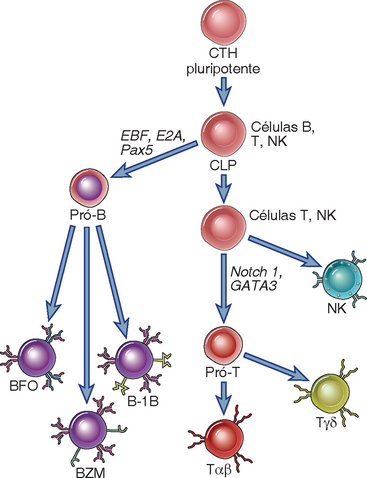
FIGURA 8-2 As células-tronco pluripotentes dão origem a linhagens B e T distintas. As células-tronco hematopoiéticas (CTH) dão origem a progenitores distintos para vários tipos de células sanguíneas. Uma dessas populações progenitoras (mostrada aqui) é denominada progenitor linfoide comum (CLT). Os CLT dão origem principalmente às células B e T, mas também podem contribuir com células NK e algumas células dendríticas (não ilustradas aqui). As células pró-B podem diferenciar-se finalmente em células B foliculares (FO), células B da zona marginal (ZM) e células B-1. As células pró-T podem ser comprometidas com as linhagens de células T αβ ou γδ. O comprometimento com a linhagem T depende de sinais liberados pela Notch-1, cujo domínio intracelular medeia a ativação de transcrição dos genes da linhagem T, em colaboração com outros fatores de transcrição, como GATA-3. O comprometimento com linhagem B é mediado, inicialmente, pelos fatores de transcrição EBF e E2A, subsequentemente, por Pax-5. Esses fatores de transcrição atuam juntos para induzir a transcrição de genes específicos das células B e de genes do mecanismo de recombinação. (Os fatores de transcrição estão indicados em itálico.)
O comprometimento com a linhagem B ou T depende de instruções recebidas de vários receptores de superfície celular, que produzem sinais para induzir reguladores específicos da transcrição que estimulam um progenitor linfoide comum a assumir especificamente o destino de uma célula B ou de uma célula T. Os receptores de superfície celular e os fatores de transcrição contribuem na indução do comprometimento através da ativação de proteínas envolvidas no rearranjo dos genes dos receptores de antígenos e tornar os loci particulares desses genes acessíveis a essas proteínas promotoras de rearranjo. No caso das células B em desenvolvimento, o lócus das cadeias pesadas de imunoglobulinas (Ig), que originalmente se encontra em uma configuração de cromatina “fechada”, é aberto, de modo a ficar acessível às proteínas que irão mediar o rearranjo e a expressão dos genes dos receptores de antígenos. Nas células T αβ em desenvolvimento, o primeiro lócus a se tornar disponível é o lócus do gene β do receptor das células T (TCR). Além daqueles envolvidos no processo de rearranjo dos genes dos receptores de antígenos, os genes que impulsionam a diferenciação subsequente das células T e B são expressos nesse estágio.
Diferentes conjuntos de fatores de transcrição norteiam o desenvolvimento das linhagens de células B e T de precursores não comprometidos (Fig. 8-2). A família Notch de proteínas são moléculas de superfície celular, proteoliticamente clivadas quando interagem com ligantes específicos em células adjacentes. As porções intracelulares clivadas das proteínas Notch migram até o núcleo e modulam a expressão de genes-alvo específicos. O Notch-1, um membro da família Notch, é ativado em células progenitoras linfoides e colabora com um fator de transcrição, denominado GATA-3, para comprometer o desenvolvimento dos linfócitos com a linhagem T. Esses reguladores da transcrição contribuem para a indução de vários genes necessários ao desenvolvimento subsequente das células T αβ. Os genes-alvo de GATA-3 incluem componentes do pré-TCR e dos mecanismos para a recombinação de V(D)J, descrito mais adiante. Nas células B, os fatores de transcrição EBF e E2A contribuem para a indução de outro fator de transcrição, denominado Pax-5, e essas três proteínas colaboram para induzir o processo de comprometimento com a linhagem B, facilitando a expressão de vários genes. Esses genes, descritos adiante de modo mais detalhado, incluem os que codificam as proteínas Rag-1 e Rag-2, as cadeias leves substitutas e as proteínas Igα e Igβ, que contribuem para a sinalização por meio do receptor da célula pré-B e receptor da célula B. O papel desses receptores no desenvolvimento da célula B será abordado mais adiante, neste capítulo.
O desenvolvimento inicial das células B e T é caracterizado pela proliferação de progenitores comprometidos, induzidos por sinais derivados de citocinas. A proliferação assegura que a geração de um reservatório de células progenitoras seja grande o suficiente para fornecer um repertório altamente diverso de linfócitos maduros específicos para antígenos. Nos roedores, a citocina interleucina-7 (IL-7) impulsiona a proliferação dos progenitores das células T e B; nos seres humanos, a IL-7 é necessária para a proliferação dos progenitores das células T, mas não para os da linhagem B. A IL-7 é produzida pelas células do estroma da medula óssea e por células epiteliais e outras células no timo. Os camundongos com mutações no gene da IL-7 ou no gene do receptor de IL-7 mostram uma maturação deficiente dos precursores dos linfócitos além dos estágios iniciais e, em consequência, apresentam deficiências profundas nas células T e B maduras. A ocorrência de mutações em uma cadeia do receptor da IL-7, denominada cadeia γ comum, pelo fato de ser compartilhada por vários receptores de citocinas, origina um distúrbio por imunodeficiência nos seres humanos que é denominado doença por imunodeficiência combinada grave ligada ao X (X-SCID). Essa doença caracteriza-se por um bloqueio no desenvolvimento das células T e das células NK, porém com desenvolvimento normal das células B, refletindo o papel da IL-7 nos seres humanos (Cap. 20).
A atividade proliferativa nos estágios iniciais do desenvolvimento dos linfócitos, impulsionada principalmente pela IL-7, cessa exatamente antes que o rearranjo dos genes para uma cadeia do receptor de antígenos seja completado; a proliferação subsequente só ocorre nas células em que houve rearranjo bem-sucedido e expressão do gene que codifica a primeira cadeia do receptor de antígenos em uma célula B ou T em desenvolvimento. Uma vez produzida essa cadeia do receptor, ela forma o receptor da célula pré-B (pré-BCR) ou o receptor da célula pré-T (pré-TCR), descritos mais adiante, que selecionam células para sobrevivência, proliferação e diferenciação subsequentes, mas que são incapazes de reconhecer antígenos. A seleção com base no pré-receptor de antígenos das células que produziram, com sucesso, uma proteína de cadeia pesada das Ig na linhagem de células B e a cadeia β do TCR na linhagem de células T αβ sustenta a maior expansão dos progenitores dos linfócitos durante o desenvolvimento. Esse é um importante ponto de controle no desenvolvimento que as células precisam processar com sucesso, visto que apenas as células que expressam o primeiro componente dos receptores de antígenos são capazes de sobreviver, se expandir e prosseguir para o próximo estágio de maturação.
MicroRNA e Desenvolvimento dos Linfócitos
Embora a expressão gênica durante o desenvolvimento dos linfócitos seja impulsionada principalmente por fatores de transcrição, um certo nível de regulação é mediado por microRNA (miRNA). Os miRNA são pequenos RNA endógenos não codificantes, inicialmente gerados no núcleo na forma de transcritos primários de miRNA mais longos que são processados nesse local por uma endorribonuclease, denominada Drosha, gerando pré-miRNA mais curtos, que possuem uma estrutura em alça e que podem ser transferidos para o citosol. No citosol, o pré-miRNA é adicionalmente processado por outra endorribonuclease, denominada Dicer, em um miRNA curto de filamento duplo com cerca de 21 a 22 pares de base de comprimento, do qual um filamento pode ser utilizado para se emparelhar com uma sequência complementar em diversos mRNA celulares. Esses mRNAs associam-se aos miRNA e a proteínas, denominadas proteínas Argonauta, para formar complexos conhecidos como RISC (complexo silenciador induzido por RNA, de RNA-induced silencing complex). Se a sequência original do miRNA de 6 a 8 pares de bases não for perfeitamente complementar ao mRNA, este último é impedido de ser traduzido de modo eficiente. Os mRNA podem ser alvos de degradação quando a complementaridade é perfeita. Em ambos os casos, o resultado é uma redução na abundância de proteínas codificadas por genes-alvo dos miRNA. Estudos de deleção gênica revelaram que os miRNA estão envolvidos no desenvolvimento das células B e T. Foi relatado que numerosos miRNAs regulam também a ativação dos linfócitos.
Rearranjo e Expressão dos Genes dos Receptores de Antígenos
O rearranjo dos genes dos receptores de antígenos é evento-chave no desenvolvimento dos linfócitos, responsável pela geração de um repertório diverso. Os produtos dos genes dos receptores de antígenos também fornecem sinais que asseguram a sobrevida seletiva dos linfócitos com especificidades úteis. Conforme discutido no Capítulo 7, cada clone dos linfócitos B ou T produz um receptor de antígenos com uma estrutura única para a ligação do antígeno. Em qualquer indivíduo, pode haver 107 ou mais clones diferentes de linfócitos B e T, cada um deles apresentando um receptor singular. A capacidade de cada indivíduo em gerar esses repertórios bastante diversos de linfócitos evoluiu de forma que não exige um número igualmente grande de genes de receptores de antígenos distintos; se não fosse assim, uma grande proporção do genoma estaria reservada para codificar o vasto número de moléculas de Ig e de TCR. Os genes dos receptores de antígenos funcionais são produzidos nas células B imaturas que se encontram na medula óssea e nas células T imaturas no timo, por meio de um processo de rearranjo gênico, desenvolvido para gerar um grande número de éxons que codificam as regiões variáveis utilizando no entanto uma fração relativamente pequena do genoma. Em qualquer linfócito em desenvolvimento, um dos numerosos segmentos gênicos das regiões variáveis é selecionado ao acaso e unido a um segmento de DNA distal. Os eventos de rearranjo do DNA que levam à produção dos receptores dos antígenos não dependem da presença ou influência de antígenos. Em outras palavras, como foi proposto pela hipótese de seleção clonal, os receptores de antígenos diversos são gerados e expressos antes do encontro com os antígenos (Fig. 1-7, Cap. 1). Os detalhes moleculares do rearranjo dos genes dos receptores de antígenos serão discutidos mais adiante, neste capítulo.
Processos de Seleção que Moldam os Repertórios de Linfócitos B e T
O processo de desenvolvimento dos linfócitos possui numerosas etapas intrínsecas, denominadas pontos de controle, nas quais as células em desenvolvimento são “testadas” e só continuam a amadurecer se a etapa precedente no processo foi concluída com sucesso. Um desses pontos de controle baseia-se na produção bem-sucedida de uma das cadeias polipeptídicas da proteína receptora de antígenos que é constituída por duas cadeias, enquanto o segundo ponto de controle exige a montagem de um receptor completo. A necessidade de passar por esses pontos de controle assegura que somente os linfócitos que completaram com sucesso os processos de rearranjo dos genes dos receptores de antígenos – e, portanto, os que tendem a ser funcionais – serão selecionados para o processo de maturação. Os processos adicionais de seleção atuam após a expressão dos receptores de antígenos e servem para eliminar os linfócitos autorreativos e potencialmente prejudiciais, bem como para comprometer as células em desenvolvimento para linhagens específicas. A seguir, será feito um resumo dos princípios gerais desses eventos de seleção.
Os pré-receptores e os receptores de antígenos liberam sinais para os linfócitos em desenvolvimento, que são necessários para a sobrevida dessas células, bem como para a sua proliferação e maturação continuada (Fig. 8-3). O rearranjo dos genes dos receptores de antígenos envolve a abertura de um lócus particular de genes de receptores (como o lócus dos genes dos TCR nas células T e o lócus de Ig nas células B) e a junção de segmentos de DNA nesse lócus para gerar um gene funcional de receptores de antígenos. Durante esse processo, bases são aleatoriamente adicionadas ou removidas entre os segmentos de genes que estão sendo unidos, maximizando, assim, a variabilidade entre os receptores. Nas células B em desenvolvimento, o primeiro gene codificante de receptores de antígenos a ter o seu rearranjo concluído é a cadeia pesada das Ig ou gene H. Nas células T αβ, a cadeia β do TCR é a primeira a ser rearranjada. As células da linhagem de linfócitos B com rearranjo bem-sucedido de seus genes da cadeia pesada de Ig expressam a proteína da cadeia Ig H e efetuam a montagem de um pré-receptor de antígenos, conhecido como pré-BCR. Da mesma forma, as células T em desenvolvimento que realizam um rearranjo produtivo dos genes da cadeia β do TCR sintetizam a proteína da cadeia β do TCR e montam um pré-receptor de antígenos, conhecido como pré-TCR. Apenas uma em três células B e T em desenvolvimento que realizam o rearranjo de um gene de receptor de antígenos faz um rearranjo adequado e, portanto, é capaz de gerar uma proteína integral apropriada. Se as células fizeram rearranjos fora do padrão adequado, nos loci da cadeia μ da Ig ou β do TCR, não ocorre expressão dos pré-receptores de antígenos, e as células que não recebem os sinais necessários de sobrevida sofrem morte celular programada. Os complexos pré-BCR e pré-TCR montados fornecem sinais para a sobrevida, para o processo de proliferação, para o fenômeno de exclusão alélico, discutido adiante, e para a continuação do desenvolvimento das células das linhagens B e T em maturação. Assim, a expressão do pré-receptor de antígenos constitui o primeiro ponto de controle durante o desenvolvimento de linfócitos.
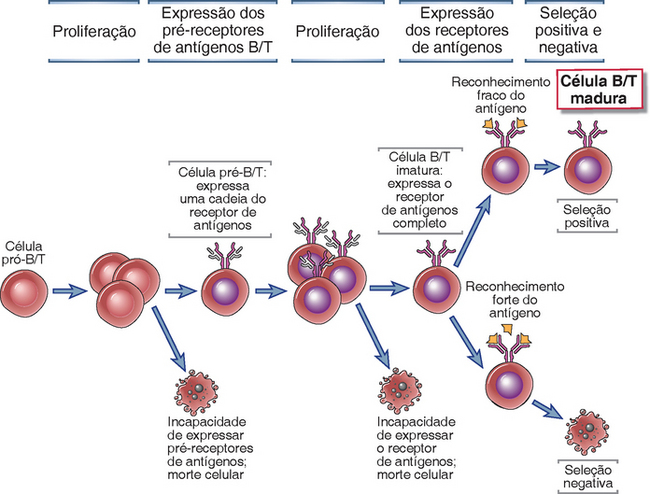
FIGURA 8-3 Pontos de controle na maturação dos linfócitos. Durante o desenvolvimento, os linfócitos que expressam receptores necessários para a continuação de sua proliferação e maturação são selecionados para sobreviver, enquanto as células que não expressam receptores funcionais morrem por apoptose. A seleção positiva e a seleção negativa preservam as células com especificidades úteis. A presença de múltiplos pontos de controle assegura que apenas as células com receptores úteis irão completar a sua maturação.
Os linfócitos que ultrapassaram com sucesso esse ponto de controle continuam seu desenvolvimento nos órgãos linfoides geradores e expressam o receptor de antígeno completo enquanto ainda estão imaturos. Nesse estágio imaturo, as células potencialmente prejudiciais, que reconhecem fortemente as estruturas próprias, podem ser eliminadas ou induzidas a alterarem os receptores de antígenos, enquanto as células que expressam receptores de antígenos úteis podem ser preservadas (Fig. 8-4). Um processo denominado seleção positiva facilita a sobrevida de linfócitos potencialmente úteis; esse evento do desenvolvimento está ligado ao comprometimento com determinada linhagem, o processo pelo qual são gerados subtipos de linfócitos. Na linhagem de células T, a seleção positiva garante a maturação das células T cujos receptores reconhecem moléculas próprias do MHC e também assegura que a expressão do correceptor apropriado na célula T (CD8 ou CD4) seja compatível com o reconhecimento do tipo apropriado de molécula do MHC (MHC da classe I ou MHC da classe II). As células T maduras, cujos precursores foram positivamente selecionados pelas moléculas próprias do MHC no timo, são capazes de reconhecer antígenos peptídicos estranhos apresentados às mesmas moléculas próprias do MHC que são expressos sobre as células apresentadoras de antígenos nos tecidos periféricos. Na linhagem de células B, a seleção positiva preserva as células que expressam receptores de antígeno e está acoplada a gerações de diferentes subgrupos, conforme discutido adiante.
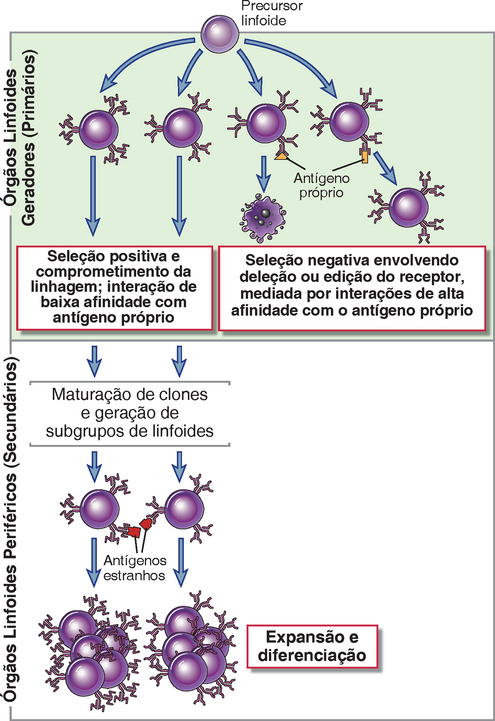
FIGURA 8-4 Seleção positiva e seleção negativa durante a maturação dos linfócitos. Após a expressão dos receptores de antígenos, os clones imaturos de linfócitos nos órgãos linfoides geradores estão sujeitos aos processos de seleção positiva e seleção negativa. Na seleção positiva, os precursores linfocíticos com receptores de antígenos que se ligam a algum ligante próprio com baixa afinidade são selecionados para sobreviver e continuar o processo de maturação. As células B em desenvolvimento recebem sinais de sobrevida simplesmente devido à expressão dos receptores de antígenos completos, sem reconhecimento de um antígeno próprio. Entretanto, como no caso das células T, o antígeno próprio de diferentes afinidades pode impulsionar a diferenciação de diferentes subgrupos de células B. Por conseguinte, a seleção positiva em ambas as linhagens de células B e T está fortemente ligada ao processo de gerar subgrupos de linfócitos, também conhecido como comprometimento com a linhagem. Os linfócitos selecionados de modo positivo entram nos tecidos linfoides periféricos, onde respondem a antígenos estranhos. Na seleção negativa, as células que se ligam com alta afinidade aos antígenos presentes nos órgãos geradores recebem sinais que levam à morte celular ou que induzem um rearranjo adicional dos genes dos receptores de antígenos, um processo conhecido como edição do receptor. Em consequência, o repertório de linfócitos maduros carece de células com capacidade de responder a esses antígenos próprios. O diagrama ilustra a seleção das células B; os princípios são os mesmos para os linfócitos T, exceto que não há edição do receptor na linhagem T.
A seleção negativa é o processo que elimina ou que altera os linfócitos em desenvolvimento, cujos receptores de antígenos ligam-se fortemente a antígenos próprios presentes nos órgãos linfoides geradores. Tanto as células B como as células T em desenvolvimento são suscetíveis à seleção negativa durante um breve período após a expressão inicial dos receptores de antígenos. As células T em desenvolvimento com alta afinidade para antígenos próprios são eliminadas por apoptose, um fenômeno conhecido como deleção clonal. As células B imaturas fortemente autorreativas podem ser induzidas a fazer rearranjos adicionais do gene das Ig, fugindo, assim, da autorreatividade. Esse fenômeno é denominado edição do receptor. Se a edição falhar, as células B autorreativas morrem, um processo também denominado deleção clonal. A seleção negativa dos linfócitos imaturos constitui um importante mecanismo na manutenção da tolerância a numerosos antígenos próprios, isso é também denominado tolerância central, visto que ocorre nos órgãos linfoides centrais (geradores) (Cap. 14).
Geração dos Subgrupos de Linfócitos
Outra característica importante do desenvolvimento dos linfócitos, ligada à seleção positiva, é a geração de subgrupos funcionalmente distintos em ambas as linhagens de células T e B. Os precursores que expressam tanto CD4 como CD8 diferenciam-se em células T CD4+ restritas ao MHC da classe II ou células T CD8+ restritas ao MHC da classe I. As células T CD4+ virgens (naïve) que deixam o timo podem ser ativadas por antígenos para se diferenciarem em células T auxiliares (helper), cujas funções efetoras são mediadas por proteínas específicas da membrana e por citocinas secretadas. As células CD8+ podem diferenciar-se em linfócitos T citotóxicos, cuja principal função efetora consiste em destruir as células-alvo infectadas. Um processo similar de comprometimento com a linhagem durante a seleção positiva das células B impulsiona o desenvolvimento dessas células em subgrupos distintos de células B na periferia. As células B em desenvolvimento, derivadas da medula óssea, podem diferenciar-se em células B foliculares, que recirculam e medeiam as respostas imunes dependentes das células T nos órgãos linfoides secundários ou podem diferenciar-se em células B da zona marginal que residem próximas ao seio marginal no baço e que medeiam, em grande parte, as respostas independentes das células T a antígenos transportados pelo sangue.
Após essa introdução, será realizada uma discussão mais detalhada da maturação dos linfócitos, começando com o evento-chave do processo, o rearranjo e a expressão dos genes dos receptores de antígenos.
REARRANJO DOS GENES DOS RECEPTORES DE ANTÍGENOS DOS LINFÓCITOS B E T
Os genes que codificam diversos receptores de antígenos dos linfócitos B e T são gerados pelo rearranjo, em cada linfócito, de diferentes segmentos gênicos presentes nas regiões variáveis (V), de diversidade (D) e de junção (J). Um novo éxon rearranjado para cada gene de um receptor de antígenos é gerado através da fusão de um dos segmentos gênicos da região V com um dos segmentos gênicos da região D e, finalmente, com um dos genes J no mesmo cromossomo. Esse processo especializado de rearranjo de genes em locais específicos é denominado recombinação V(D)J. (Os termos recombinação e rearranjo são empregados como sinônimos.) A elucidação dos mecanismos de rearranjo dos genes dos receptores de antígenos e, portanto, da base da geração da diversidade imunológica representa uma das principais conquistas da imunologia moderna.
As primeiras noções de como milhões de diferentes receptores de antígenos podiam ser gerados a partir de uma quantidade limitada de DNA no genoma provêm de análises das sequências de aminoácidos das moléculas de Ig. Essas análises mostraram que as cadeias polipeptídicas de muitos anticorpos diferentes do mesmo isótipo compartilhavam sequências idênticas em suas extremidades C-terminais (correspondendo aos domínios constantes das cadeias pesadas do anticorpo), porém diferiam consideravelmente nas sequências das extremidades N-terminais, que correspondem aos domínios variáveis das imunoglobulinas (Cap. 5). Ao contrário de um dos dogmas centrais da genética molecular, conhecido como a “hipótese de um gene-um polipeptídio” por Beadle e Tatum, em 1941. Dreyer e Bennett postularam, em 1965, que cada cadeia de anticorpo é, na realidade, codificada por pelo menos dois genes, um variável e outro constante, e que os dois se combinam fisicamente em nível do DNA ou do RNA mensageiro (mRNA) para por fim dar origem às proteínas de Ig funcionais. A prova formal dessa hipótese apareceu mais de uma década depois, quando Susumu Tonegawa demonstrou que a estrutura dos genes das Ig nas células de um tumor produtor de anticorpos, denominado mieloma ou plasmocitoma, é diferente daquela presente nos tecidos embrionários ou nos tecidos não linfoides que não são comprometidos com a produção de Ig. Essas diferenças surgem porque os segmentos de DNA nos loci que codificam as cadeias pesadas e leves da Ig estão especificamente ligados entre si apenas nas células B em desenvolvimento, mas não em outros tecidos ou tipos de células. Foi constatada a ocorrência de rearranjos semelhantes durante o desenvolvimento das células T nos loci que codificam as cadeias polipeptídicas dos TCR. É mais fácil compreender o rearranjo dos genes dos receptores de antígenos descrevendo, inicialmente, a organização não rearranjada dos genes das Ig e dos TCR na linhagem germinativa, e, em seguida, o seu rearranjo durante a maturação dos linfócitos.
Organização dos Genes da Ig e do TCR nas Linhagens Germinativas
A organização dos loci genéticos das Ig e dos TCR na linhagem germinativa é fundamentalmente semelhante e caracteriza-se pela segregação espacial de múltiplas sequências, que codificam domínios variáveis e constantes das proteínas receptoras; as sequências das regiões variáveis distintas são unidas a sequências das regiões constantes em diferentes linfócitos. Serão descritos, em primeiro lugar, os loci da Ig e, em seguida, os dos TCR.
Organização dos Loci Gênicos das Ig
Três loci separados codificam, respectivamente, todas as cadeias pesadas e as cadeias leves κ e λ das Ig. Cada lócus encontra-se em um cromossomo diferente. A Figura 8-5 ilustra a organização dos genes das Ig humanas, enquanto a relação dos segmentos gênicos, após o rearranjo, com os domínios das proteínas das cadeias pesadas e leves das Ig é mostrada na Figura 8-6A. Os genes das Ig estão organizados essencialmente da mesma forma em todos os mamíferos, embora a sua localização cromossômica e o número e a sequência dos diferentes segmentos gênicos em cada lócus possam variar.
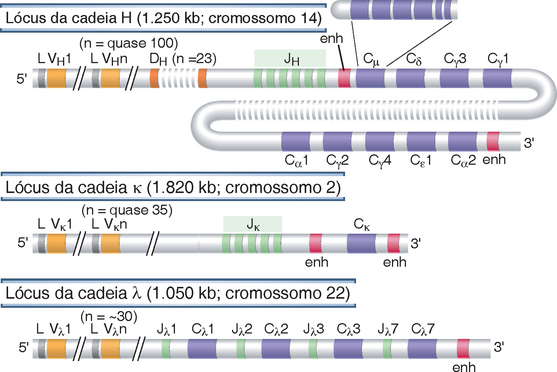
FIGURA 8-5 Organização dos loci das Ig humanas na linhagem germinativa. São mostrados os loci das cadeias pesadas e das cadeias leves κ e λ humanas. Apenas os genes funcionais são mostrados; os pseudogenes foram omitidos para maior simplicidade. Os éxons e os íntrons não estão representados na escala. Cada gene CH é representado como único quadrado, porém é composto de vários éxons, conforme ilustrado para Cμ. Os segmentos gênicos estão indicados da seguinte forma: L, líder (frequentemente denominado sequência de sinal); V, variável; D, diversidade; J, junção; C, constante; enh, amplificador.
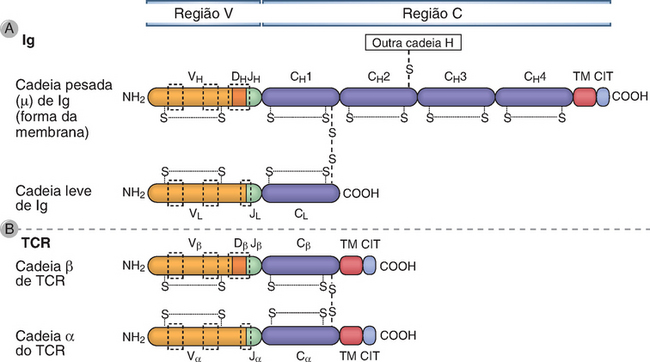
FIGURA 8-6 Domínios das proteínas Ig e TCR. Em A, são mostrados os domínios das cadeias pesada e leve das Ig, e, em B, os domínios das cadeias α e β do TCR. As relações entre os segmentos gênicos das Ig e TCR e a estrutura dos domínios das cadeias polipeptídicas do receptor de antígenos estão indicadas. As regiões V e C de cada polipeptídio são codificadas por diferentes segmentos gênicos. As localizações das ligações de dissulfeto (S-S) intracadeia e entre cadeias são aproximadas. As áreas nos retângulos tracejados representam as regiões hipervariáveis (que determinam a complementaridade). Na cadeia μ das Ig e nas cadeias α e β dos TCRs, os domínios transmembrana (TM) e citoplasmático (CIT) são codificados por éxons separados.
Na extremidade 5′ de cada um dos loci das Ig, existe um segmentos gênico que contém diferentes genes chamados V, e cada gene V possui cerca de 300 pares de bases. O número de genes V varia consideravelmente entre os diferentes loci das Ig e entre espécies diferentes. Por exemplo, existem cerca de 35 genes V no lócus da cadeia leve κ humana, cerca de 30 no lócus λ e cerca de 100 genes V funcionais no lócus da cadeia pesada humana; enquanto o lócus da cadeia leve λ do rato tem apenas dois genes V e o lócus da cadeia pesada do rato possui mais de 1.000 genes V. Os segmentos gênicos V em cada lócus estão espaçados por intervalos longos de DNA, de até 2.000 quilobases de comprimento. Na extremidade 5′ de cada segmento V, existe um éxon líder, que codifica de 20 a 30 resíduos N-terminais na proteína traduzida. Esses resíduos são moderadamente hidrofóbicos e formam o peptídio líder (ou sinal). São encontradas sequências de sinais em todas as proteínas secretadas e transmembrana recém-sintetizada, que estão envolvidas na orientação dos polipeptídios nascentes traduzidos nos ribossomos ligados à membrana no lúmen do retículo endoplasmático. No retículo endoplasmático, as sequências sinais são rapidamente clivadas e não estão presentes nas proteínas maduras. Próximo a cada éxon líder, encontra-se um promotor do gene V onde a transcrição pode ser iniciada; entretanto, conforme discutido adiante, isso ocorre de modo mais eficiente após o rearranjo.
Em distâncias variáveis de 3′ dos genes V, encontram-se vários segmentos J estreitamente ligados aos éxons que codificam a região constante, que são encontrados mais distalmente. Os segmentos J têm, normalmente, um comprimento de 30 a 50 pares de base e são separados por sequências não codificadoras. Entre os segmentos V e J no lócus IgH, existem segmentos adicionais, conhecidos como segmentos D. À semelhança dos genes V, o número de genes J e D varia em diferentes loci das Ig e em espécies diferentes.
Cada lócus de Ig possui o arranjo distinto e número variados de genes que codificam a região C. Nos seres humanos, o lócus da cadeia leve κ possui um único gene C (Cκ), e o lócus da cadeia leve λ possui quatro genes C (Cλ) funcionais. O lócus da cadeia pesada das Ig tem nove genes C (CH), dispostos em fileira, que codificam as regiões C dos nove isótipos diferentes e subgrupos de Ig (Cap. 5). Cada gene Cκ e Cλ é composto de um único éxon, que codifica todo o domínio C das cadeias leves. Por sua vez, cada gene CH é constituído de cinco ou seis éxons. Três ou quatro éxons (cada um deles com tamanho semelhante a um segmento gênico V) codificam, cada um deles, um domínio CH da cadeia pesada das Ig, enquanto dois éxons menores codificam as extremidades carboxiterminais da forma associada à membrana de cada cadeia pesada das Ig, incluindo os domínios transmembrana e citoplasmático das cadeias pesadas (Fig. 8-6A).
A proteína das cadeias leves de Ig (κ ou λ), o domínio V é codificado pelos segmentos gênicos V e J; a proteína das cadeias pesadas de Ig, o domínio V é codificado pelos segmentos gênicos V, D e J (Fig. 8-6A). Todos os resíduos de junção entre os segmentos V e D e os segmentos D e J rearranjados, bem como a sequência dos próprios segmentos D e J, formam a terceira região hipervariável (também conhecida como região determinante da complementaridade 3 ou CDR3) no caso dos domínios V de IgH e β do TCR. As sequências juncionais entre os segmentos V e J rearranjados, bem como o próprio segmento J, constituem a terceira região hipervariável das cadeias leves de Ig. Os CDR1 e CDR2 são codificados no próprio segmento gênico V da linhagem germinativa. Os domínios V e C das moléculas de Ig compartilham características estruturais, incluindo uma estrutura terciária, denominada dobra da Ig. Conforme discutido no Capítulo 5, as proteínas que incluem essa estrutura são membros da superfamília das Ig.
As sequências não codificadoras nos loci das Ig desempenham papéis importantes na recombinação e na expressão dos genes. Conforme discutido mais adiante, as sequências que ditam a recombinação de diferentes segmentos gênicos são encontradas adjacentes a cada segmento codificador nos genes de Ig. Existem também promotores do gene V e outros elementos reguladores de atuação cis, como as regiões de controle do lócus, amplificadores e silenciadores, que regulam a expressão gênica em nível da transcrição.
Organização dos Loci dos Genes dos TCR
Os genes que codificam as cadeias α, β e γ dos TCR estão mapeados em três loci distintos em três cromossomos diferentes, enquanto o lócus da cadeia δ dos TCR encontra-se dentro do lócus α do TCR (Fig. 8-7). Cada lócus de TCR na linhagem germinativa inclui segmentos gênicos V e J, estando este último exatamente proximal aos éxons da região C em cada lócus. Além disso, os loci TCR β e TCR δ também possuem segmentos D, como o lócus das cadeias pesadas de Ig. Na extremidade 5′ de cada um dos loci dos TCR, existe um grupamento de vários segmentos gênicos V, dispostos de forma muito semelhante aos segmentos gênicos V das Ig. Numa posição proximal a cada gene V de TCR, há um éxon que codifica um peptídio líder, e, adjacente a cada éxon líder, existe um promotor para cada gene V.
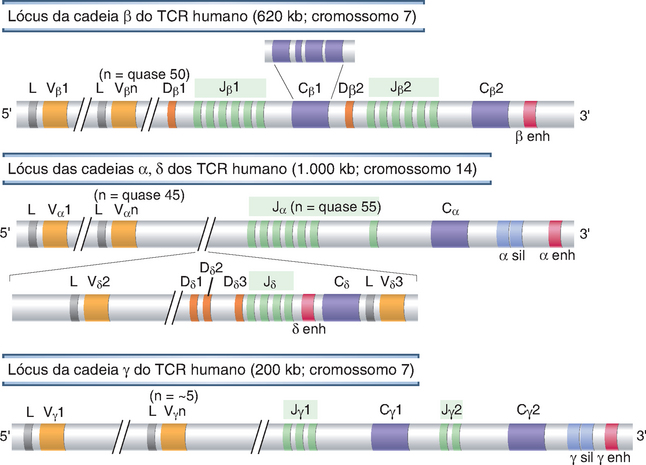
FIGURA 8-7 Organização dos loci dos TCR humanos na linhagem germinativa. São mostrados os loci das cadeias β, α, γ e δ dos TCR humanos. Os éxons e os íntrons não estão representados na escala, e os pseudogenes não funcionais não estão ilustrados. Cada gene C é mostrado como um único retângulo, porém é composto de vários éxons, como ilustrado para Cβ1. Os segmentos gênicos estão indicados da seguinte forma: L, líder (habitualmente denominada sequência de sinal); V, variável; D, diversidade; J, junção; C, constante; enh, amplificador; sil, silenciador (sequências que regulam a transcrição dos genes dos TCR).
Em cada lócus de TCR, à semelhança dos loci das Ig, os genes da região C estão localizados exatamente em 3′ dos segmentos J. Existem dois genes C em cada um dos loci de TCR β (Cβ) e TCR γ (Cγ) humanos, e apenas um gene C em cada um dos loci TCR α (Cα) e TCR δ (Cδ). Cada gene da região C do TCR é composto de quatro éxons, que codificam o domínio semelhante à região C extracelular das Ig, uma região de dobradiça curta, o segmento transmembrana e a cauda citoplasmática. Os loci das cadeias β e δ dos TCR assemelham-se ao lócus das cadeias pesadas de Ig e contêm segmentos D entre os genes V e os segmentos J. Cada gene C do TCR humano encontra-se imediatamente a 3′ dos segmentos J. Nas cadeias α ou γ dos TCR (análogas às cadeias leves das Ig), o domínio V é codificado pelos éxons V e J; ao passo que, nas proteínas β e δ do TCR, o domínio V é codificado pelos segmentos gênicos V, D e J.
A Figura 8-6B mostra a relação dos segmentos gênicos dos TCR e as porções correspondentes das proteínas do TCR que elas codificam. Como nas moléculas de Ig, os domínios V e C dos TCR assumem uma estrutura terciária contendo dobras típicas das Ig, e, por conseguinte, o TCR é um membro da superfamília de proteínas da Ig.
Recombinação V(D)J
A organização dos loci das Ig e dos TCR na linha germinativa, descrita na sessão anterior, é encontrada em todos os tipos de células no corpo. A organização dos genes encontrados na linhagem germinativa impede que estes possam ser transcritos em mRNA, que, consequentemente, não podem codificar proteínas receptoras funcionais de antígenos. Os genes que codificam os receptores funcionais de antígenos são apenas criados nos linfócitos B e T durante o seu desenvolvimento após os eventos de rearranjo do DNA, que estabelecem uma continuidade dos segmentos gênicos V, (D) e J aleatoriamente escolhidos. A Figura 8-8 mostra, de modo esquemático, um Southern blot, incluindo um lócus de cadeia leve das Ig na configuração da linhagem germinativa em uma célula não linfoide. O segmento V mostra nesta configuração uma distância considerável dos segmentos J descritos. Um rearranjo hipotético no clone da célula B é também mostrado. Neste clone, um segmento específico V em direção vertical foi adicionado a um dos segmentos J durante o processo de rearranjo.
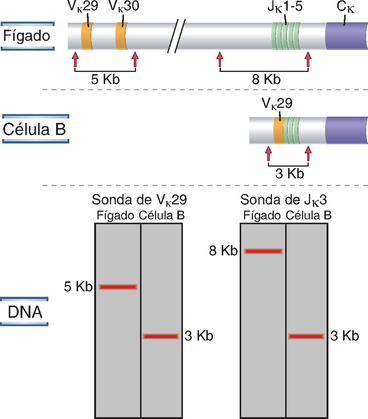
FIGURA 8-8 Rearranjos dos genes dos receptores de antígenos. A análise Southern blot do DNA de células não linfoides (do fígado) e de uma população monoclonal de origem de linhagem de linfócitos B (p. ex., tumor de células B) é mostrada de modo esquemático. O DNA é digerido por uma enzima de restrição (EcoRI, como ilustrado), os fragmentos de tamanhos diferentes são separados por eletroforese e transferidos para um filtro. Os locais em que a enzima de restrição EcoRI cliva o DNA estão indicados por setas. O tamanho dos fragmentos contendo o segmento Jκ3 do gene da cadeia leve κ das Ig ou gene da região Vκ29 foi determinado mediante o uso de uma sonda radioativa que se liga especificamente ao segmento Jκ3 do DNA ou ao Vκ29 do DNA. No exemplo hipotético apresentado, Vκ29 faz parte de um fragmento de EcoRI de 5 kb nas células hepáticas, porém está em um fragmento de 3 kb no clone de células B estudado. De forma semelhante, o fragmento Jκ3 tem 8 kb nas células hepáticas, porém 3 kb no clone de células B.
O processo de recombinação V(D)J em qualquer lócus de Ig ou TCR envolve a seleção de um gene V, um gene J e um dos genes do segmento D (quando presente) em cada linfócito, e o rearranjo desses segmentos gênicos em conjunto forma o único éxon V(D)J que irá codificar a região variável de uma proteína do receptor de antígenos (Fig. 8-9). Nos loci das cadeias leves de Ig e das cadeias α e γ dos TCR, que carecem de segmentos D, um único evento de rearranjo une um gene V aleatoriamente selecionado a um gene do segmento J, também selecionado de modo aleatório. Os loci IgH e TCR β e δ contêm segmentos D, e, nesses loci, dois eventos distintos de rearranjo devem ser iniciados separadamente: o primeiro unindo um gene do segmento D com um gene do segmento J e, então, um dos genes do segmento V que é escolhidos e unido ao segmento DJ previamente rearranjado. Cada evento de rearranjo envolve várias etapas sequenciais. Primeiro, a cromatina deve ser aberta em regiões específicas do cromossomo do receptor de antígenos para que os segmentos gênicos fiquem acessíveis às enzimas que medeiam a recombinação. Em seguida, dois segmentos gênicos selecionados, que se encontram numa distância considerável no cromossoma, precisam ser aproximados um do outro. Em seguida, são introduzidas quebras na dupla fita do DNA em regiões próximas às extremidades codificadoras desses dois segmentos e nucleotídeos são adicionados ou removidos extremidades quebradas, e, por fim, as extremidades processadas são ligadas formando assim genes funcionais capazes de codificar receptores de antígenos únicos que são expressos na superfície de clone de linfócito. Devido a esses mecanismos de recombinação, diferentes receptores são gerados contra diversos antígenos. As regiões C estão localizadas distalmente ao éxon V(D)J rearranjado, separado pelo íntron J-C da linhagem germinativa. Esse éxon rearranjado é transcrito para formar um transcrito de RNA primário (nuclear). O processamento (splicing) subsequente do RNA aproxima o éxon líder, o éxon V(D)J e os éxons da região C, formando um mRNA que pode ser traduzido em cadeias do receptor de antígenos. O uso de diferentes combinações dos segmentos gênicos V, D e J e a adição e remoção de nucleotídios nas junções contribuem para a enorme diversidade dos receptores de antígenos, como será discutido de modo mais detalhado adiante.
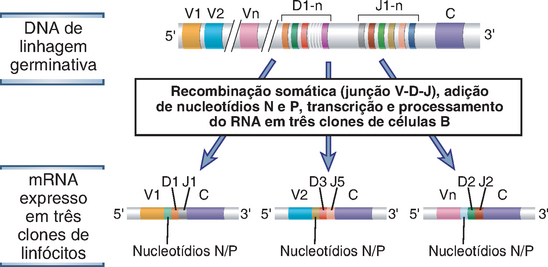
FIGURA 8-9 Diversidade dos genes dos receptores de antígenos. Do DNA da mesma linhagem germinativa, é possível gerar sequências recombinadas de DNA e de mRNA, que diferem em suas junções V-D-J. No exemplo apresentado, são produzidos três mRNA de receptores de antígenos distintos do mesmo DNA de linhagem germinativa, utilizando segmentos gênicos diferentes e adição de nucleotídios nas junções.
Sinais de Reconhecimento que Impulsionam a Recombinação V(D)J
Fatores críticos específicos dos linfócitos, que medeiam a recombinação V(D)J, reconhecem determinadas sequências de DNA, denominadas sequências sinais de recombinação (RSS), localizadas em 3′ de cada segmento gênico V, 5′ de cada segmento J e ambos os lados de cada segmento D (Fig. 8-10A). Os RSS consistem em um segmento altamente conservado de sete nucleotídios, denominado heptâmero, normalmente CACAGTG, de localização adjacente à sequência codificadora, seguida de um espaçador contendo exatamente 12 ou 23 nucleotídios não conservados, seguido de um segmento altamente conservado de nove nucleotídios, rico em AT, denominado nanômero. Os espaçadores de 12 e de 23 nucleotídios correspondem a cerca de uma ou duas voltas da hélice de DNA, respectivamente, e presume-se que eles colocam dois heptâmeros distintos em posições simultaneamente acessíveis às enzimas que catalisam o processo de recombinação.
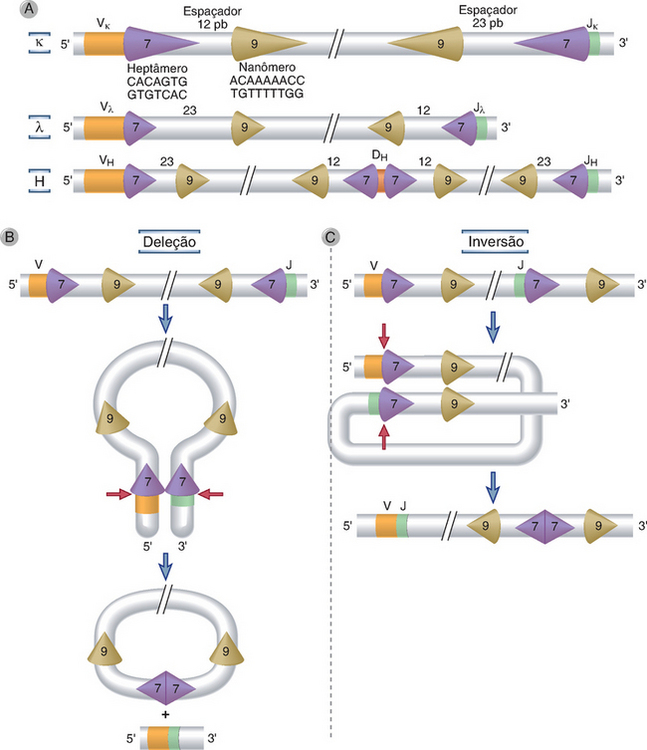
FIGURA 8-10 Recombinação V(D)J. São mostradas as sequências de DNA e os mecanismos envolvidos na recombinação dos loci dos genes das Ig. As mesmas sequências e mecanismos aplicam-se à recombinação dos loci dos TCR. A, As sequências de heptâmero (7 pb) e nonâmero (9 pb) conservadas, separadas por espaçadores de 12 ou 23 pb, estão adjacentes aos éxons V e J (para os loci κ e λ) ou aos éxons V, D e J (no lócus da cadeia H). A V(D)J recombinase reconhece essas sequências de sinais de recombinação e aproxima os éxons. B, C. A recombinação dos éxons V e J pode ocorrer por deleção do DNA entre eles e pela ligação dos segmentos V e J (B) ou, se o gene V estiver na orientação oposta, pela inversão do DNA, seguida de ligação dos segmentos gênicos adjacentes (C). As setas vermelhas indicam os locais onde as sequências das linhagens germinativas são clivadas antes de sua ligação a outros segmentos gênicos das Ig ou dos TCR.
Durante a recombinação V(D)J, são produzidas quebras de filamento duplo entre o heptâmero da RSS e a sequência adjacente que codifica V, D e J. Por exemplo, na recombinação V-para-J da cadeia leve das Ig, são feitas quebras em 3′ de um segmento V e 5′ de um segmento J. O DNA de filamento duplo interveniente, que contém as extremidades de sinais (as extremidades que contêm o heptâmero e o resto da RSS), é removido na forma de um círculo, e esse processo é acompanhado da junção das extremidades que codificam V e J (Fig. 8-10B). Alguns genes V, em particular, no lócus κ das Ig, estão na mesma orientação que os segmentos J, de modo que o RSS em 5′ desses segmentos V e 3′ dos segmentos J não estão na “frente” um do outro. Nesses casos, o DNA interveniente é invertido, os éxons V e J são alinhados apropriadamente e as RSS fundidas não sofrem deleção, porém são retidas no cromossomo (Fig. 8-10C). A maior parte dos rearranjos dos genes das Ig e dos TCR ocorre por deleção; ocorre rearranjo por inversão em até 50% dos rearranjos no lócus κ. A recombinação entre dois segmentos só ocorre se um dos segmentos estiver ladeado por um espaçador de 12 nucleotídios e se o outro estiver ladeado por um espaçador de 23 nucleotídios, constituindo a denominada regra 12/23. Por conseguinte, um segmento codificador com uma RSS de “uma volta” sempre se combina com um segmento codificador com uma RSS de “duas voltas”. O tipo de RSS que ladeia (uma volta ou duas voltas) assegura a recombinação dos segmentos gênicos apropriados. Por exemplo, no lócus da cadeia pesada das Ig, as RSS que ladeiam ambos os segmentos V e J apresentam espaçadores de 23 nucleotídios (duas voltas) e, por conseguinte, não podem unir-se diretamente; primeiro, ocorre recombinação D-para-J, seguida de recombinação de V-para-DJ, e isso é possível pelo fato de que os segmentos D são cercados, em ambos os lados, por espaçadores de 12 nucleotídios, permitindo a junção D-J e então V-DJ. As RSS descritas aqui são exclusivas dos genes das Ig e dos TCR. Por conseguinte, a recombinação V(D)J pode ocorrer nos genes dos receptores de antígenos, mas não em outros genes.
Uma das consequências da recombinação V(D)J é que o processo aproxima promotores, localizados imediatamente em 5′ dos genes V, com amplificadores distais, que estão localizados nos íntrons J-C e também em 3′ dos genes da região C (Fig. 8-11). Esses amplificadores maximizam a atividade de transcrição dos promotores do gene V e, portanto, são importantes para a transcrição de alto nível dos genes V rearranjados nos linfócitos. Como os genes das Ig e dos TCR constituem locais de múltiplos eventos de recombinação do DNA nas células B e T e como esses locais tornam-se ativos para transcrição após a recombinação, os genes de outros loci podem ser anormalmente translocados para esses loci e, em consequência, podem ser transcritos de modo anormal. Nos tumores de linfócitos B e T, os oncogenes são, com frequência, translocados para os loci dos genes das Ig ou dos TCR. Essas translocações cromossômicas são, com frequência, acompanhadas de aumento da transcrição dos oncogenes, e acredita-se que seja um dos fatores que causam o desenvolvimento de tumores linfoides.
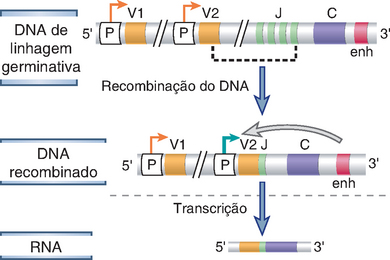
FIGURA 8-11 Regulação da transcrição dos genes Ig. A recombinação V-D-J aproxima as sequências promotoras (ilustradas como P) do amplificador (enh). O amplificador promove a transcrição do gene V rearranjado (V2, cujo promotor ativo está indicado por uma seta verde). Muitos genes de receptores possuem um amplificador no íntron J-C e outro 3′ da região C. Apenas o amplificador 3′ está ilustrado aqui.
O Mecanismo de Recombinação V(D)J
O rearranjo dos genes das Ig e dos TCR representa um tipo especial de recombinação não homóloga do DNA, mediado pelas atividades coordenadas de várias enzimas, algumas das quais são encontradas apenas nos linfócitos em desenvolvimento, enquanto outras são enzimas ubíquas de reparo de quebras de filamento duplo do DNA (DSBR). Embora o mecanismo de recombinação V(D)J esteja bastante elucidado e seja descrito aqui, ainda não foi estabelecido como exatamente os loci específicos tornam-se acessíveis ao processo envolvido na recombinação. É provável que a acessibilidade dos loci das Ig e dos TCR às enzimas que medeiam a recombinação seja regulada, nas células B e T em desenvolvimento, por diversos mecanismos, incluindo alterações na estrutura da cromatina, metilação do DNA e atividade de transcrição basal nos loci gênicos.
O processo de recombinação V(D)J pode ser dividido em quatro eventos distintos, que seguem um fluxo sequencial (Fig. 8-12):
1. Sinapse: Porções do cromossomo no qual está localizado o gene do receptor de antígenos que tornam-se acessíveis ao processo de recombinação. Dois segmentos codificadores selecionados e suas RSS adjacentes são aproximados por um evento formador de uma alça no cromossomo, sendo mantidos nessa posição para clivagem, processamento e junção subsequentes.
2. Clivagem: Quebras de filamento duplo são produzidas enzimaticamente nas junções das sequências codificadoras de RSS por um mecanismo que é específico das células linfoides. Duas proteínas codificadas por genes linfoides específicos, denominadas gene ativador da recombinação 1 e gene ativador da recombinação 2 (Rag-1 e Rag-2), formam um complexo tetramérico que desempenha papel essencial na recombinação V(D)J. O complexo Rag-1/Rag-2 é também conhecido como V(D)J recombinase. A proteína Rag-1, de forma semelhante a uma endonuclease de restrição, reconhece a sequência de DNA na junção entre um heptâmero e um segmento codificador, clivando-o, porém só é ativa enzimaticamente quando complexada com a proteína Rag-2. A proteína Rag-2 pode ajudar a ligar o tetrâmero Rag-1/Rag-2 a outras proteínas, incluindo fatores de acessibilidade, que levam essas proteínas até loci gênicos de receptores “abertos” específicos, em momentos determinados e em estágios definidos do desenvolvimento dos linfócitos. A Rag-1 e a Rag-2 contribuem para manter unidos os segmentos gênicos durante o processo de dobramento ou sinapse do cromossomo. Em, seguida, a Rag-1 faz um corte (em um filamento) entre a extremidade codificadora e o heptâmero. O terminal 3′ OH, liberado da extremidade codificadora, ataca então uma ligação fosfodiéster no outro filamento, formando uma estrutura covalente semelhante a um grampo. A extremidade sinal (incluindo o heptâmero e o restante da RSS) não forma uma estrutura em forma de grampo e é gerada como uma terminação romba de DNA de filamento duplo, que não é mais processada. Essa quebra de filamento duplo resulta em um grampo fechado de um segmento codificador mantido em aposição ao grampo fechado da outra extremidade codificadora e duas extremidades de sinais rombas de recombinação são colocadas uma ao lado da outra. Além de produzir as quebras de filamento duplo, a Rag-1 e a Rag-2 também mantêm as extremidades dos grampos e as extremidades rombas unidas antes da modificação das extremidades codificadoras e do processo de ligação.
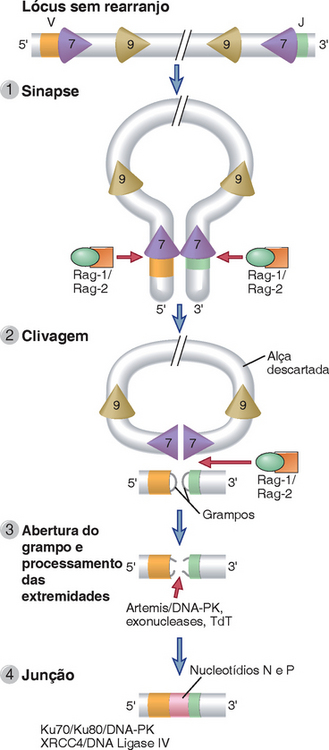
FIGURA 8-12 Eventos sequenciais durante a recombinação V(D)J. A sinapse e a clivagem do DNA no limite heptâmero/segmento codificador são mediadas pela Rag-1 e Rag-2. O grampo na extremidade codificadora é aberto pela endonuclease Artemis, e ocorre reparo das extremidades quebradas pelo mecanismo NHEJ.
Os genes Rag são específicos dos linfócitos e são expressos apenas nas células B e T em desenvolvimento. As proteínas Rag são expressas principalmente nos estágios G0 e G1 do ciclo celular e são inativadas nas células em proliferação. Acredita-se que a limitação da clivagem e da recombinação do DNA a esses estágios minimiza o risco de gerar quebras inapropriadas do DNA durante a sua replicação ou durante a mitose.
3. Abertura do grampo e processamento das extremidades: As extremidades codificadoras quebradas (mas não as extremidades sinais/RSS) são modificadas pela adição ou remoção de bases, com consequente geração de uma maior diversidade. Após a formação de quebras de filamento duplo, os grampos devem ser abertos nas junções codificadoras, e as bases podem ser adicionadas ou removidas das extremidades codificadoras para assegurar uma diversificação ainda maior. A Artemis é uma endonuclease que abre os grampos nas extremidades codificadoras. Na ausência de Artemis, os grampos não podem ser abertos e não pode haver geração de células T e B maduras. Uma imunodeficiência rara, caracterizada pela ausência de célula T e B, é causada por mutações no Artemis (Cap. 20). Uma enzima específica do tecido linfoide, denominada desoxinucleotidil transferase terminal (TdT), acrescenta bases às extremidades quebradas do DNA pela ação da Artemis e é discutida posteriormente, neste capítulo, no contexto da diversidade juncional.
4. Junção: As extremidades codificadoras quebradas, bem como as extremidades de sinais, são aproximadas e ligadas por um processo de reparo de quebras de filamento duplo encontrado em todas as células, denominado junção terminal não homóloga. Diversos fatores ubíquos participam da junção terminal não homóloga. A Ku70 e a Ku80 são proteínas que ligam as extremidades do DNA quebradas e assim recrutam a subunidade catalítica da proteinocinase dependente de DNA (DNA-PK), uma enzima de reparo do DNA de filamento duplo. Essa enzima está deficiente em camundongos portadores da mutação que causa imunodeficiência combinada grave (scid) (Cap. 20). Assim como os camundongos deficientes em Rag, os camundongos com scid são incapazes de produzir linfócitos maduros. A DNA-PK também fosforila e ativa a Artemis, que, conforme mencionado, está envolvida no processamento das extremidades. A ligação das extremidades quebradas processadas é mediada pela DNA ligase IV e pela XRCC4, sendo esta última uma subunidade não catalítica, porém essencial dessa ligase.
Geração de Diversidade nas Células B e T
A enorme diversidade dos repertórios de células B e T é criada não apenas por combinações aleatórias de segmentos gênicos de linhagem germinativa reunidos, como também pela adição ou deleção aleatórias de sequências nas junções entre os segmentos unidos. Diversos mecanismos genéticos contribuem para essa diversidade, e a importância relativa de cada mecanismo varia entre os diferentes loci dos receptores de antígenos (Tabela 8-1). A hipermutação somática, um mecanismo que envolve mutações pontuais e outras alterações no DNA nas células B ativadas, não é discutida aqui e será considerada no Capítulo 11.
TABELA 8-1 Contribuições de Diferentes Mecanismos na Geração da Diversidade nos Genes da Ig e dos TCR
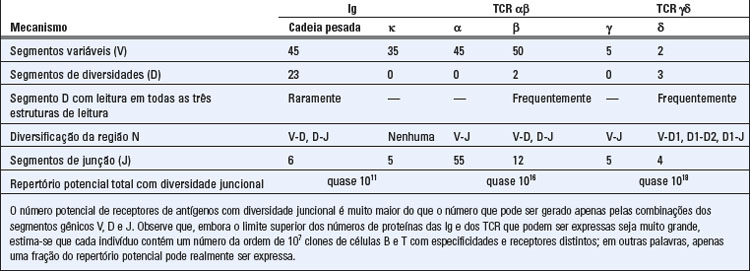
• Diversidade combinatória. O rearranjo V(D)J reúne múltiplos segmentos gênicos de linhagem germinativa, que podem combinar-se de modo aleatório, e diferentes combinações produzem receptores de antígenos distintos. O número máximo possível de combinações desses segmentos gênicos é o produto do número de segmentos gênicos V, J e (quando presente) D em cada lócus de receptor de antígenos. Por conseguinte, a quantidade de diversidade combinatória que pode ser gerada em cada lócus reflete o número de segmentos gênicos V, J e D de linhagem germinativa naquele lócus. Após a síntese das proteínas dos receptores de antígenos, a diversidade combinatória é ainda maior devido à justaposição de duas regiões V diferentes (i. e., VH e VL nas moléculas de Ig e Vα e Vβ nas moléculas de TCR) que são geradas de modo aleatório). Por conseguinte, a diversidade combinatória total é, teoricamente, o produto da diversidade combinatória de cada uma das duas cadeias associadas. O grau real de diversidade combinatória nos repertórios de Ig e TCR expressos em qualquer indivíduo tende a ser consideravelmente menor do que o máximo teórico. Isso se deve ao fato de que nem todas as recombinações de segmentos gênicos têm a mesma probabilidade de ocorrer, e nem todos os pares de cadeias pesadas e leves das Ig ou das cadeias α e β dos TCR podem formar receptores de antígenos funcionais. O aspecto mais importante é que, como o número de segmentos V, D e J em cada lócus é limitado (Tabela 8-1), o número máximo possível de combinações é de milhares. Esse número é, obviamente, muito menor do que a diversidade real dos receptores de antígenos nos linfócitos maduros.
• Diversidade juncional. A maior contribuição à diversidade dos receptores de antígenos é feita pela remoção ou adição de nucleotídios nas junções dos segmentos V e D, D e J ou V e J por ocasião da junção desses segmentos. Uma forma pela qual isso pode ocorrer é por meio das endonucleases, que removem nucleotídios das sequências de linhagem germinativa nas extremidades dos segmentos gênicos recombinantes. Além disso, novas sequências de nucleotídios, não presentes na linhagem germinativa, podem ser adicionadas às junções. Conforme descrito anteriormente, os segmentos codificadores (p. ex., segmentos gênicos V e J) clivados pela Rag-1 formam alças em forma de grampo, cujas extremidades são, com frequência, clivadas assimetricamente pela enzima Artemis, de modo que um filamento de DNA é mais longo do que o outro (Fig. 8-13). O filamento mais curto precisa ser alongado com nucleotídios complementares ao filamento mais longo antes da ligação dos dois segmentos. Os nucleotídios adicionados aos filamentos curtos são denominados nucleotídios P, e a sua adição por modelo introduz novas sequências nas junções V-D-J. Outro mecanismo de diversidade juncional é a adição aleatória de até 20 nucleotídios sem uso de um molde, denominados nucleotídios N (Fig. 8-3). A diversificação da região N é mais comum nas cadeias pesadas das Ig e nas cadeias β e γ dos TCR do que nas cadeias κ ou λ das Ig. Essa adição de novos nucleotídios é mediada pela enzima desoxinucleotidil transferase terminal (TdT). Em camundongos com deficiência induzida de TdT por nocautear os genes para TdT, a diversidade dos repertórios de células B e T é substancialmente menor do que nos camundongos normais. A adição de nucleotídios P e N nos locais de recombinação pode introduzir alterações na estrutura (frameshifts), gerando, teoricamente, códons de terminação em dois de cada três eventos de junção. Esses genes não podem produzir proteínas funcionais, porém essa ineficiência é o preço que se paga pela geração da diversidade.
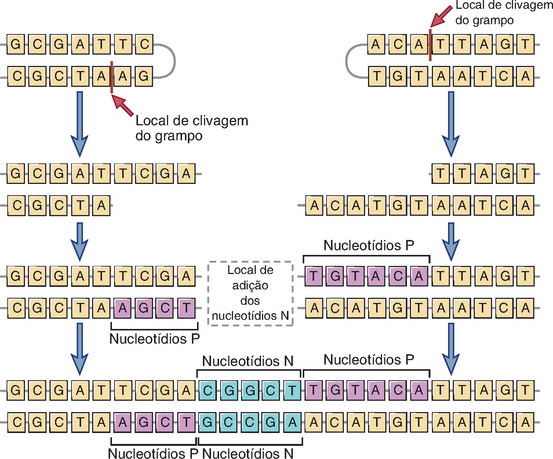
FIGURA 8-13 Diversidade juncional. Durante a junção de diferentes segmentos gênicos, a adição ou a remoção de nucleotídios pode levar à geração de novas sequências de nucleotídios e aminoácidos na junção. Os nucleotídios (sequências P) podem ser adicionados a grampos clivados de modo assimétrico de uma forma modelada. Outros nucleotídios (regiões N) podem ser adicionados aos locais das junções VD, VJ ou DJ, de forma não modelada (sem uso de um molde) pela ação da enzima TdT. Essas adições geram novas sequências que não estão presentes na linhagem germinativa.
Devido à diversidade juncional, as moléculas de anticorpos e de TCR exibem a maior variabilidade nas junções das regiões V e C, que formam a terceira região hipervariável ou CDR3 (Fig. 8-6). De fato, devido à diversidade juncional, os números de sequências diferentes de aminoácidos presentes nas regiões CDR3 das moléculas de Ig e de TCR são muito maiores do que o número que pode ser codificado por segmentos gênicos da linhagem germinativa. As regiões CDR3 das moléculas de Ig e de TCR também constituem as porções mais importantes dessas moléculas para determinar a especificidade de ligação dos antígenos (Caps. 5 e 7). Assim, a diversidade máxima nos receptores de antígenos está concentrada nas regiões dos receptores mais importantes para a ligação do antígeno.
Embora o limite teórico do número de proteínas de Ig e de TCR que podem ser produzidas seja enorme (Tabela 8-1), o número verdadeiro de receptores de antígenos expressos nas células B ou T de cada indivíduo é, provavelmente, da ordem de apenas 107. Isso pode refletir o fato de que a maioria dos receptores, gerados por recombinação aleatória do DNA, não passa pelos processos de seleção necessários para a maturação.
Uma aplicação prática do conhecimento sobre a diversidade juncional consiste na determinação da clonalidade dos tumores linfoides que se originam de células B ou T. Esse exame laboratorial é comumente utilizado para diferenciar os tumores monoclonais da proliferação policlonal dos linfócitos em resposta a algum estímulo externo. Como cada clone de linfócitos expressa uma região CDR3 do receptor de antígenos única, a sequência de nucleotídios no local de recombinação V(D)J serve como marcador específico de cada clone. Assim, ao medir o comprimento das regiões juncionais dos genes das Ig ou dos TCR em diferentes células B ou T por ensaios da reação em cadeia da polimerase, pode-se estabelecer se essas sequências se originam de um único clone (indicando um tumor) ou por clones diferentes (indicando uma proliferação não neoplásica de linfócitos). O mesmo método pode ser utilizado para identificar pequenos números de células tumorais no sangue ou nos tecidos.
Com essa base, pode-se prosseguir na discussão do desenvolvimento dos linfócitos B e, em seguida, na maturação das células T.
DESENVOLVIMENTO DOS LINFÓCITOS B
Os principais eventos que ocorrem durante a maturação dos linfócitos B consistem no rearranjo e na expressão dos genes das Ig em uma ordem precisa, na seleção e na proliferação das células B em desenvolvimento, no ponto de controle dos pré-receptores de antígenos e na seleção do repertório de células B maduras. Antes do nascimento, os linfócitos B desenvolvem-se de precursores comprometidos no fígado do feto, e, depois do nascimento, as células B são geradas na medula óssea. Os linfócitos B surgem, em sua maioria, de progenitores na medula óssea do adulto que, inicialmente, são negativos para as Ig, desenvolvem-se em células B imaturas que expressam moléculas de IgM ligadas à membrana e, em seguida, deixam a medula óssea para amadurecer principalmente no baço. No baço, as células que se desenvolvem em células B foliculares que expressam IgM e IgD em sua superfície e adquirem a capacidade de recircular e ocupar todos os órgãos linfoides periféricos. Essas células B foliculares dirigem-se para os folículos linfoides, onde são capazes de reconhecer antígenos estranhos e responder a eles. Estima-se que o desenvolvimento de uma célula B madura de um progenitor linfoide leve 2 a 3 dias nos seres humanos.
Estágios de Desenvolvimento dos Linfócitos B
Durante a sua maturação, as células da linhagem dos linfócitos B passam por estágios específicos, cada um caracterizado por marcadores distintos de superfície celular e por um padrão específico de expressão dos genes das Ig (Fig. 8-14). Em seguida, são descritos os principais estágios e eventos em cada um deles.
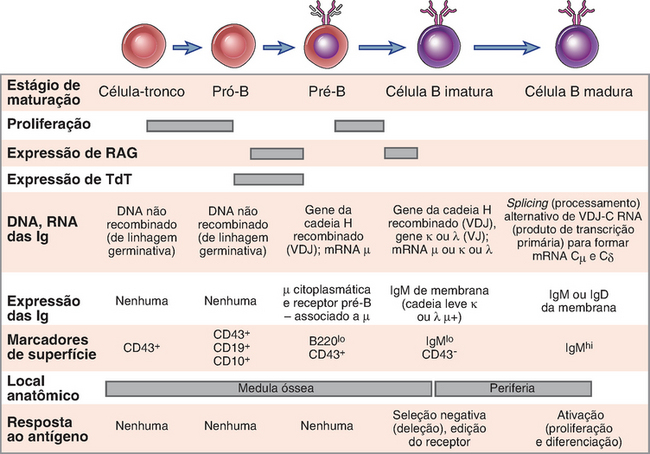
FIGURA 8-14 Estágios de maturação de células B. São ilustrados os eventos que correspondem a cada estágio de maturação de células B, da célula-tronco na medula óssea até o linfócito B maduro. Foram utilizados vários marcadores de superfície, além dos mostrados, para definir os estágios distintos de maturação das células B.
Estágios Pró-B e Pré-B de Desenvolvimento das Células B
A primeira célula da medula óssea comprometida com a linhagem de células B é denominada célula pró-B. As células pró-B não produzem Ig, mas podem ser distinguidas de outras células imaturas pela expressão de moléculas de superfície restritas à linhagem B, como CD19 e CD10. As proteínas Rag são inicialmente expressas nesse estágio, e a primeira recombinação dos genes das Ig ocorre no lócus das cadeias pesadas. Essa recombinação aproxima um segmento gênico D e um segmento gênico J, com deleção do DNA intercalado entre eles (Fig. 8-15A). Os segmentos D que estão em 5′ do segmento D rearranjado e os segmentos J que estão em 3′ do segmento J rearranjado não são afetados por essa recombinação (p. ex., D1 e J2 para J6 na Fig. 8-15A). Após a ocorrência da recombinação D-J, um dos numerosos genes V 5′ une-se à unidade DJ, dando origem a um éxon VDJ rearranjado. Nesse estágio, todos os segmentos V e D entre os genes V e D rearranjados também sofrem deleção. A recombinação V-para-DJ no lócus da cadeia H das Ig só ocorre nos precursores comprometidos dos linfócitos B e constitui um evento crítico na expressão das Ig, visto que apenas o gene V rearranjado é subsequentemente transcrito. A enzima TdT, que catalisa a adição sem modelo dos nucleotídios N juncionais, é expressa de modo mais abundante durante o estágio pró-B, quando ocorre a recombinação VDJ no lócus Ig H, e os níveis de TdT diminuem antes de completar a recombinação V-J dos genes das cadeias leves. Por isso, a diversidade juncional atribuída à adição de nucleotídios N é mais proeminente nos genes rearranjados das cadeias pesadas do que nos genes das cadeias leves. Os éxons da região C das cadeias pesadas permanecem separados do complexo VDJ por DNA contendo os segmentos J distais e o íntron J-C. O gene das cadeias pesadas de Ig rearranjado é transcrito para produzir um produto de transcrição primária que inclui o complexo VDJ rearranjado e os éxons Cμ. O RNA nuclear Cμ é clivado distalmente a um de dois locais de poliadenilação de consenso, e múltiplos nucleotídios de adenina, denominados caudas de poli-A, são adicionados à extremidade 3′. Esse RNA nuclear sofre maturação (splicing), um evento no processamento do RNA, em que os íntrons são removidos, e os éxons são unidos. No caso do RNAμ, os íntrons entre o éxon líder e o éxon VDJ, entre o éxon VDJ e o primeiro éxon do lócus Cμ e entre cada um dos éxons subsequentes da região constante de Cμ, são removidos dando origem, assim, a um mRNA cuja a porção constante da cadeia pesada é do tipo μ (IgM). Se o mRNA for derivado de um lócus de Ig, em que o arranjo foi produtivo, a tradução do mRNA da cadeia pesada μ rearranjado leva à síntese da proteína μ. Para que um rearranjo seja produtivo (na fase de leitura correta), é preciso que haja adição ou remoção de bases nas junções em múltiplos de três. Isso assegura que o gene das Ig rearranjado seja capaz de codificar de forma correta uma proteína de Ig. Cerca da metade de todas as células pró-B realiza rearranjos produtivos no lócus H da Ig em pelo menos um cromossomo e, portanto, pode sintetizar a proteína da cadeia pesada μ. Apenas as células que fazem rearranjos produtivos sobrevivem e continuam a se diferenciar.
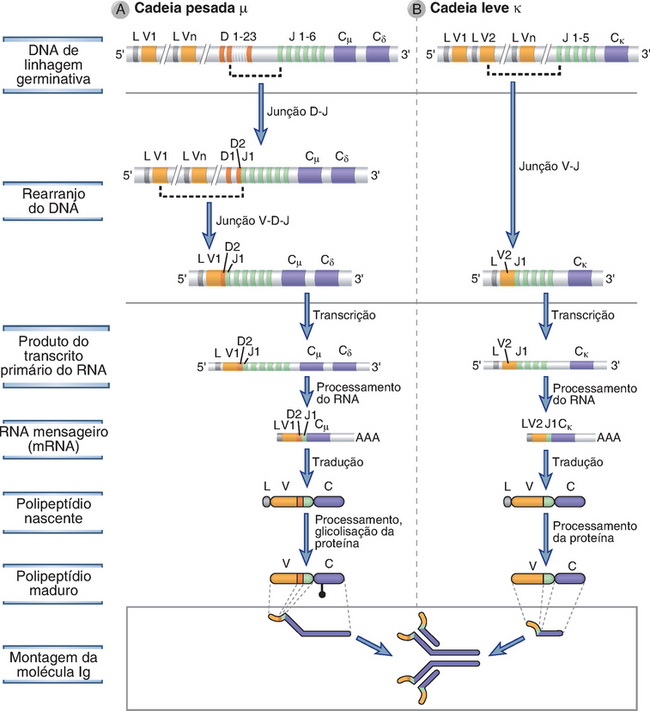
FIGURA 8-15 Recombinação e expressão dos genes das cadeias pesadas e leves das Ig. São mostrados a sequência de recombinação do DNA e os eventos de expressão dos genes para a cadeia pesada μ (A) e a cadeia leve κ (B) das Ig. No exemplo apresentado em A, a região V da cadeia pesada μ é codificada pelos éxons V1, D2 e J1. No exemplo mostrado em B, a região V da cadeia κ é codificada pelos éxons V2 e J1.
Uma vez realizado um rearranjo μ de Ig produtivo, a célula não é mais denominada célula pró-B e já está diferenciada no estágio pré-B. As células pré-B são células de linhagem B em desenvolvimento, que expressam a proteína μ das Ig, mas que ainda precisam rearranjar os loci das cadeias leves. A célula pré-B expressa a cadeia pesada μ na superfície celular, em associação a outras proteínas, como o pré-receptor de células B, que desempenha várias funções importantes na maturação das células B.
O Receptor de Células Pré-B
O pré-receptor de antígenos da linhagem B, conhecido como receptor da célula pré-B (pré-BCR), é formado por complexos de cadeias leves substitutas, de μ e por proteínas de transdução de sinais, denominadas Ig α e Ig β. A cadeia pesada μ associa-se às proteínas λ5 e pré-B V, também denominadas cadeias leves substitutas, visto que são estruturalmente homólogas às cadeias leves κ e λ, porém não apresentam variação (i. e., são idênticas em todas as células pré-B) e são sintetizadas apenas nas células pró-B e pré-B (Fig. 8-16A). A Igα e a Igβ também fazem parte do receptor de células B nas células B maduras (Cap. 7). Os sinais do pré-BCR impulsionam a transição de pró-B para pré-B e são responsáveis pela maior expansão proliferativa das células de linhagem B na medula óssea. Não se sabe o que e o pré-BCR reconhece; na atualidade, o ponto de vista consensual é de que esse receptor atua de forma independente do ligante, sendo ativado pelo processo de montagem (i. e., o receptor, após a sua montagem completa, encontra-se no modo “ligado”). A importância dos pré-BCRs é ilustrada por estudos de camundongos nocauteados (knockouts) e por raros casos de deficiência desses receptores em seres humanos. Por exemplo, nos camundongos, a deficiência do gene que codifica a cadeia μ, ou que codifica uma das cadeias leves substitutas, resulta em uma acentuada redução no número de células B maduras, visto que o desenvolvimento é bloqueado no estágio pró-B.
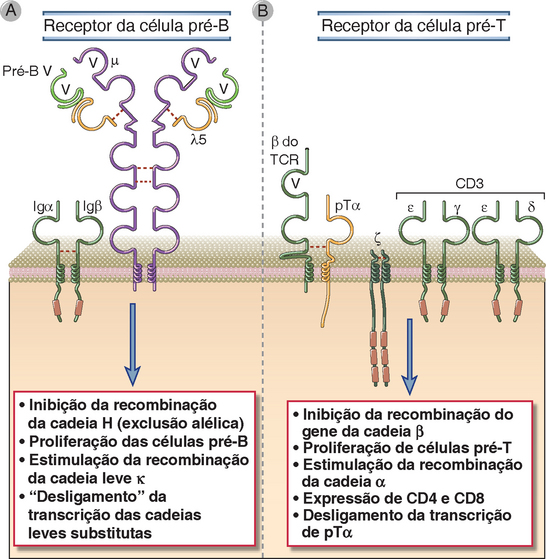
FIGURA 8-16 Receptores das células pré-B e pré-T. O receptor da célula pré-B (A) e o receptor da célula pré-T (B) são expressos durante os estágios de maturação das células pré-B e pré-T, respectivamente, e ambos os receptores compartilham estruturas e funções similares. O receptor da célula pré-B é composto da cadeia pesada μ e de uma cadeia leve substituta invariável. A cadeia leve substituta é composta de duas proteínas, a proteína V pré-B, que é homóloga ao domínio V da cadeia leve, e a proteína λ5, que está ligada de modo covalente à cadeia pesada μ por uma ligação de dissulfeto. O receptor da célula pré-T é composto da cadeia β do TCR e da cadeia α pré-T (pTα) invariável. O receptor de células pré-B está associado às moléculas de sinalização Igα e Igβ, que fazem parte do complexo do BCR nas células B maduras (Cap. 9), enquanto o receptor de células pré-T está associado às proteínas CD3 e ζ, que fazem parte do complexo do TCR nas células T maduras (Cap. 7).
A expressão do pré-BCR constitui o primeiro ponto de controle na maturação das células B. São necessárias numerosas moléculas de sinalização ligadas ao pré-BCR e aos BCRs para que as células ultrapassem, com sucesso, o ponto de controle mediado pelo pré-BCR na transição de pró-B para pré-B. Uma cinase, denominada tirosinocinase de Bruton (Btk), é ativada distalmente ao pré-BCR e é necessária para transmitir sinais desse receptor, que medeiam a sobrevida, a proliferação e a maturação no estágio de célula pré-B e além dele. Nos seres humanos, a ocorrência de mutações no gene BTK provoca uma doença denominada agamaglobulinemia ligada ao X (XLA), caracterizada por uma incapacidade de maturação das células B (Cap. 20). Nos camundongos, as mutações de btk resultam em um defeito menos grave das células B em uma cepa de camundongos denominada Xid (imunodeficiência ligada ao X, do inglês, X-linked immunodeficiency). O defeito é menos grave do que na XLA, visto que as células pré-B murinas expressam uma segunda cinase semelhante à Btk, denominada Tec, que compensa a Btk deficiente.
O pré-BCR regula o rearranjo adicional dos genes das Ig de duas formas. Na primeira, se uma proteína μ for produzida do lócus recombinado das cadeias pesadas em um cromossomo e formar um pré-BCR, esse receptor emite um sinal para inibir irreversivelmente o rearranjo do lócus das cadeias pesadas de Ig no outro cromossomo. Se o primeiro rearranjo não for produtivo, o alelo das cadeias pesadas no outro cromossomo pode completar o rearranjo VDJ no lócus H das Ig. Assim, em qualquer clone de células B, um alelo da cadeia pesada sofre rearranjo produtivo e é expresso, enquanto o outro é retido na configuração da linhagem germinativa ou sofre rearranjo não produtivo. Como resultado, uma célula pode expressar proteínas das cadeias pesadas de Ig codificadas por apenas um dos dois alelos herdados. Esse fenômeno é denominado exclusão alélica e assegura que cada célula B irá expressar um único receptor, mantendo, assim, a especificidade clonal. Se ambos os alelos sofrem rearranjos não produtivos do gene H das Ig, a célula em desenvolvimento é incapaz de produzir cadeias pesadas de Ig, não pode gerar um sinal de sobrevida dependente do pré-BCR e, portanto, sofre morte celular programada. A exclusão alélica das cadeias pesadas de Ig envolve alterações na estrutura da cromatina no lócus das cadeias pesadas, limitando a acessibilidade à V(D)J recombinase.
A segunda forma pela qual o pré-BCR regula a produção do receptor de antígenos consiste na estimulação do rearranjo do gene da cadeia leve κ. Todavia, a expressão da cadeia μ não é absolutamente necessária para a recombinação do gene da cadeia leve, conforme demonstrado pelo achado de que camundongos nocautes (knockouts), que carecem do gene μ, iniciam rearranjos do gene da cadeia leve em algumas células B em desenvolvimento (que, naturalmente, não podem expressar receptores de antígenos funcionais e prosseguir no processo de maturação). O pré-BCR também contribui para a inativação da expressão dos genes de cadeias leves substitutas à medida que as células pré-B amadurecem.
Células B Imaturas
Após o estágio de célula pré-B, cada célula B em desenvolvimento rearranja inicialmente um gene da cadeia leve κ, e, se o rearranjo for produtivo, irá gerar uma proteína da cadeia leve κ, que se associa à cadeia μ previamente sintetizada para produzir uma proteína IgM completa. Se não houver rearranjo produtivo do lócus κ, a célula pode rearranjar o lócus λ e novamente produzir uma molécula IgM completa. (A indução do rearranjo do gene da cadeia leve λ ocorre principalmente quando os receptores das células B que expressam κ da Ig são autorreativos, conforme discutido adiante.) A célula B que expressa IgM é denominada célula B imatura. A recombinação do DNA no lócus da cadeia leve κ ocorre de modo semelhante à do lócus das cadeias pesadas de Ig (Fig. 8-15B). Não há segmentos D nos loci das cadeias leves, e, por conseguinte, a recombinação envolve apenas a junção de um segmento V com um segmento J, formando um éxon VJ. Esse éxon VJ permanece separado da região C por um íntron, e essa separação é retida na transcrição primária do RNA. O processamento (splicing) do produto de transcrição primária leva à remoção do íntron entre os éxons VJ e C e gera um mRNA, que é traduzido para produzir a proteína κ ou λ. No lócus λ, o processamento (splicing) alternativo do RNA pode levar ao uso de qualquer um dos quatro éxons Cλ funcionais, porém não existe nenhuma diferença funcional conhecida entre os tipos resultantes de cadeias leves λ. A produção de uma proteína κ impede o rearranjo λ, e, conforme mencionado, o rearranjo λ só ocorre se o rearranjo κ não for produtivo ou se ocorrer deleção de uma cadeia leve κ rearranjada autorreativa. Como resultado, um clone de células B só pode expressar um dos dois tipos de cadeias leves; esse fenômeno é denominado exclusão do isótipo de cadeia leve. Como no caso do lócus da cadeia pesada, a expressão de κ ou λ sofre exclusão alélica e só é iniciada em um dos dois cromossomos herdados em qualquer momento determinado. Além disso, como no caso das cadeias pesadas, se ambos os alelos para as cadeias κ e λ não forem funcionais em uma célula B em desenvolvimento, essa célula não recebe sinais de sobrevida normalmente gerados pelo BCR e, consequentemente, morre.
As moléculas de IgM montadas são expressas na superfície celular, em associação a Igα e Igβ, onde funcionam como receptores específicos de antígenos. Nas células não fortemente autorreativas, o BCR fornece sinais centrais que são independentes de ligante, mas que mantêm a célula B viva ao mesmo tempo que inibe a expressão do gene Rag, impedindo, assim, qualquer rearranjo adicional dos genes das Ig. As células B imaturas não proliferam e não se diferenciam em resposta a antígenos. De fato, quando reconhecem antígenos na medula óssea com alta afinidade, o que pode ocorrer se as células B expressam receptores para antígenos próprios multivalentes presentes na medula óssea, as células B podem sofrer edição do receptor ou morte celular, conforme descrito adiante. Esses processos são importantes para a seleção negativa das células B fortemente autorreativas. As células B imaturas que não são fortemente autorreativas deixam a medula óssea e completam a sua maturação no baço, antes de migrar para outros órgãos linfoides periféricos.
Subgrupos de Células B Maduras
Subgrupos distintos de células B desenvolvem-se de diferentes progenitores (Fig. 8-17). As CTH derivadas do fígado fetal são os precursores das células B-1, descritas adiante. As CTH derivadas da medula óssea dão origem à maior parte das células B, algumas vezes denominadas células B-2. Essas células passam rapidamente por dois estágios de transição e podem se comprometer com o desenvolvimento de células B da zona marginal, também descrito adiante, ou células B foliculares. A afinidade do receptor de células B para antígenos próprios contribui para a diferenciação, em uma célula B folicular ou uma célula B da zona marginal, de uma célula B-2 em maturação. Essa decisão quanto ao destino das células representa um evento de seleção positiva nos linfócitos B, ligado ao comprometimento da linhagem.
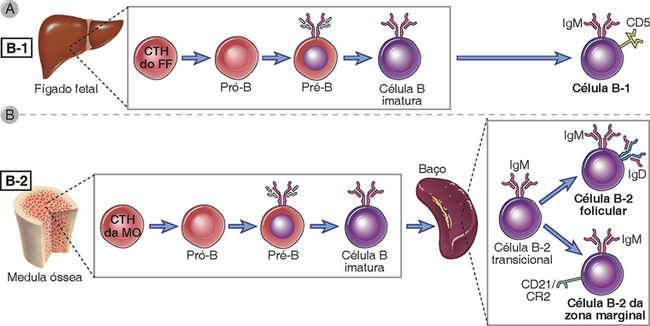
FIGURA 8-17 Subgrupos de linfócitos B. A, As células B que se desenvolvem das células-tronco derivadas do fígado fetal diferenciam-se, em sua maioria, na linhagem B-1. B, Os linfócitos B que surgem de precursores da medula óssea após o nascimento dão origem à linhagem B-2. Dois subgrupos importantes de linfócitos B derivam dos precursores da célula B-2. As células B foliculares são linfócitos recirculantes, enquanto as células B da zona marginal são abundantes no baço de roedores, mas também podem ser encontradas nos gânglios linfáticos dos seres humanos.
Células B Foliculares
As células B maduras pertencem, em sua maioria, ao subgrupo de células B foliculares e expressam IgD além das IgM. Cada uma dessas células B coexpressa cadeias pesadas μ e δ que utilizam o mesmo éxon VDJ para gerar o domínio V e se associam à mesma cadeia leve, κ ou λ, para produzir dois receptores de membrana com a mesma especificidade antigênica. A expressão simultânea em uma única célula B que expressa um éxon VDJ rearranjado de especificidade única em dois produtos de transcrição, um deles incluindo éxons Cμ e o outro incluindo éxons Cδ, é obtida por processamento (splicing) alternativo do RNA (Fig. 8-18). O transcrito de RNA é longo e contém a unidade VDJ rearranjada, bem como os genes Cμ e Cδ. Se o produto de transcrição primário for clivado e poliadenilado após os éxons μ, os íntrons são retirados, de modo que o éxon VDJ fique contíguo aos éxons Cμ; isso leva à geração de um mRNA μ. Entretanto, se o complexo VDJ não estiver ligado a éxons Cμ, porém unido a éxons Cδ, ocorre produção de um mRNA δ. A tradução subsequente resulta na síntese de uma proteína completa da cadeia pesada μ ou δ. Assim, a poliadenilação seletiva e o processamento (splicing) alternativo permitem a uma célula B produzir simultaneamente mRNA maduros e proteínas de dois isótipos diferentes de cadeias pesadas. Estão pouco elucidados os mecanismos precisos que regulam a escolha da poliadenilação ou locais aceptores de união por meio dos quais o VDJ rearranjado é unido a Cμ ou Cδ, assim como os sinais que determinam quando e por que uma célula B expressa tanto IgM como IgD, e não apenas IgM. A coexpressão de IgM e de IgD é acompanhada da capacidade de recircular e da aquisição de competência funcional, e esta é a razão pela qual as células B IgM+IgD+ são também denominadas células B maduras. Essa correlação entre a expressão de IgD e a aquisição de competência funcional levou à sugestão de que a IgD constitui o receptor de ativação essencial das células B maduras. Entretanto, não há evidências de uma diferença funcional entre a IgM e a IgD de membrana. Além disso, deficiência no gene δ das Ig em camundongos não apresenta um impacto significativo sobre a maturação ou as respostas induzidas por antígenos das células B. Com frequência, as células B foliculares são também denominadas células B recirculantes, visto que elas migram de um órgão linfoide para o outro, residindo em focos especializados, conhecidos como folículos de células B (Cap. 2). Nesses nichos, as células B são mantidas, em parte, por sinais de sobrevivência emitidos por uma citocina da família do fator de necrose tumoral (TNF), denominada BAFF ou BlyS (Cap. 11).
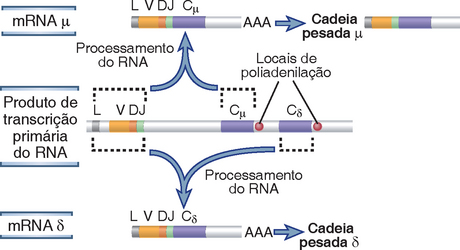
FIGURA 8-18 Coexpressão da IgM e da IgD. O processamento alternativo de um produto de transcrição primário do RNA leva à formação de um mRNA μ ou δ. As linhas tracejadas indicam os segmentos da cadeia H unidos por splicing (processamento) do RNA.
As células B maduras virgens são responsivas aos antígenos, e, a não ser que as células encontrem antígenos que reconhecem com alta afinidade e respondem a eles, elas morrem em poucos meses. No Capítulo 11, será descrito como essas células respondem aos antígenos e como o padrão de expressão dos genes da Ig muda durante a diferenciação das células B induzidas por antígenos.
Células B-1 e Células B da Zona Marginal
Um subgrupo de linfócitos B, denominado células B-1, difere da maioria dos linfócitos B e desenvolve-se de forma singular. Essas células desenvolvem-se de CTHs derivadas do fígado fetal e são mais bem definidas nos roedores. As células B-1 murinas expressam, em sua maioria, a molécula CD5 (Ly-1). No adulto, são encontrados grandes números de células B-1 como uma população autorrenovável no peritônio e em locais de mucosa. As células B-1 desenvolvem-se mais cedo durante a ontogenia do que as células B convencionais, expressam um repertório relativamente limitado de genes V e exibem uma diversidade juncional bem menor do que a das células B convencionais (visto que a TdT não é expressa no fígado fetal). As células B-1, bem como as células B da zona marginal, secretam espontaneamente anticorpos IgM que, com frequência, reagem com polissacarídios e lipídios microbianos. Algumas vezes, esses anticorpos são denominados anticorpos naturais, visto que estão presentes nos indivíduos sem imunização evidente, embora seja possível que a flora microbiana do intestino constitua a fonte de antígenos que estimula a sua produção. As células B-1 contribuem para a rápida produção de anticorpos contra micro-organismos em determinados tecidos, como o peritônio. Nas mucosas, até metade das células secretoras de IgA na lâmina própria pode ter a sua origem das células B-1. As células B-1 são análogas às células T γδ, visto que ambas possuem repertórios de receptores de antígenos de diversidade limitada, e acredita-se que ambas respondam a antígenos microbianos comumente encontrados no início da resposta imune.
O principal marcador utilizado para delinear as células B-1 murinas é o CD5. Nos seres humanos, foram descritas células semelhantes à B-1; todavia, essas células não expressam CD5. Nos seres humanos, a molécula CD5 é encontrada nas células B transicionais e em algumas populações de células B ativadas.
As células B da zona marginal estão localizadas principalmente nas proximidades do seio marginal do baço e assemelham-se às células B-1 quanto à sua diversidade limitada e à capacidade de responder a antígenos polissacarídios e de produzir anticorpos naturais. As células B da zona marginal existem tanto nos camundongos como nos seres humanos e expressam a IgM e o correceptor CD21. Nos camundongos, as células B da zona marginal só existem no baço, ao passo que, nos seres humanos, podem ser encontradas no baço e nos gânglios linfáticos. As células B da zona marginal respondem rapidamente a micro-organismos transportados pelo sangue e diferenciam-se em plasmócitos secretores de IgM de vida curta. Apesar de geralmente mediarem as respostas imunes humorais independentes das células T contra patógenos circulantes, as células B da zona marginal também parecem ser capazes de mediar algumas respostas imunes dependentes das células T.
Seleção do Repertório de Células B Maduras
O repertório das células B maduras é selecionado positivamente do reservatório de células B imaturas. Como será visto adiante, a seleção positiva está bem definida nos linfócitos T e é responsável pela compatibilidade dos TCR em células CD8+ e CD4+ recém-geradas com a sua capacidade de reconhecer moléculas próprias do MHC da classe I e da classe II, respectivamente. Não existe nenhuma restrição comparável para o reconhecimento de antígenos pela célula B. Entretanto, a seleção positiva parece ser um fenômeno geral, adaptado principalmente para a identificação dos linfócitos que completaram, com sucesso, o seu programa de rearranjo dos genes dos receptores de antígenos. Acredita-se que apenas as células B que expressam moléculas Ig funcionais da membrana recebam sinais de sobrevida constitutivos derivados do BCR, também conhecidos como sinais “tônicos” de BCR. Os sinais tônicos de BCR medeiam o desligamento da expressão dos genes Rag nas células B imaturas e ativam vias de sobrevida celular em todas as células B. Os antígenos próprios podem influenciar a intensidade do sinal do BCR e, portanto, a escolha subsequente da linhagem de células B periférica durante a maturação das células B.
As células B imaturas que reconhecem os antígenos próprios com alta afinidade podem ser induzidas a mudar suas especificidades por um processo denominado edição do receptor. Nesse processo, o reconhecimento dos antígenos leva à reativação dos genes Rag permitindo assim eventos adicionais de recombinação entre V-J das cadeias leves e à produção de uma nova cadeia leve de Ig, permitindo a expressão, pela célula, de um receptor de células B diferente, que não é autorreativo. Em geral, a edição do receptor é dirigida para os genes da cadeia leve κ autorreativa. Os éxons VJκ que codificam os domínios variáveis das cadeias leves autorreativas sofrem deleção e são substituídos por novos éxons VJκ ou por genes de cadeia leve λ rearranjados. O novo éxon VJκ pode ser gerado pelo rearranjo de um gene Vκ que se encontra próximo ao gene Vκ original que produziu uma cadeia leve autorreativa para um segmento Jκ distal ao segmento Jκ originalmente rearranjado.
Se a edição do receptor falhar, as células B imaturas, que expressam receptores de alta afinidade para antígenos próprios e que encontram esses antígenos na medula óssea ou no baço, podem morrer por apoptose. Esse processo é denominado seleção negativa. Os antígenos que medeiam a seleção negativa – normalmente antígenos próprios abundantes ou polivalentes (p. ex., ligados à membrana) – emitem fortes sinais para os linfócitos B imaturos que expressam IgM, que também expressam receptores específicos para esses antígenos próprios. A edição do receptor e a deleção são responsáveis pela manutenção da tolerância das células B aos antígenos próprios presentes na medula óssea (Cap. 14).
Uma vez feita a transição para o estágio de célula B madura IgD+IgM+, o reconhecimento do antígeno leva à proliferação e diferenciação, e não à edição do receptor ou à apoptose. Como resultado, as células B maduras que reconhecem antígenos com alta afinidade nos tecidos linfoides periféricos são ativadas, e esse processo leva às respostas imunes humorais. As células B foliculares produzem a maior parte das respostas humorais dependentes de células T auxiliares a antígenos proteicos (Cap. 11).
MATURAÇÃO DOS LINFÓCITOS T
A maturação dos linfócitos T a partir dos progenitores comprometidos envolve o rearranjo sequencial e a expressão dos genes de TCR, a proliferação celular, a seleção induzida por antígenos e o comprometimento com subgrupos fenotípica e funcionalmente distintos (Fig. 8-19). Em muitos aspectos, esse processo assemelha-se à maturação das células B. Entretanto, a maturação das células T exibe algumas características singulares, que refletem a especificidade da maioria dos linfócitos T por antígenos peptídicos próprios associados ao MHC e a necessidade de um microambiente especial para selecionar células com essa especificidade.
FIGURA 8-19 Estágios de maturação das células T. São ilustrados os eventos que correspondem a cada estágio de maturação da célula T, desde a célula-tronco na medula óssea até o linfócito T maduro. Vários marcadores de superfície, além dos mostrados, são utilizados para definir os estágios distintos de maturação da célula T.
Papel do Timo na Maturação das Células T
O timo constitui o principal local de maturação das células T. Essa função do timo foi inicialmente sugerida devido à associação de deficiências imunológicas na ausência do timo. A ausência congênita do timo, como a que ocorre na síndrome de DiGeorge nos seres humanos ou na cepa do camundongo nude, caracteriza-se por baixos números de células T maduras na circulação e nos tecidos linfoides periféricos e por grave deficiência na imunidade mediada por células T (Cap. 20). Se o timo for removido de um camundongo no período neonatal, esse animal não desenvolve células T maduras. O primórdio do timo desenvolve-se do endoderma da terceira bolsa faríngea e mesênquima derivado da crista neural subjacente e, subsequentemente, é ocupado por precursores derivados da medula óssea. O timo involui com a idade e praticamente não é detectável nos seres humanos após a puberdade, resultando em produção ligeiramente reduzida de células T maduras. Entretanto, a maturação das células T continua durante toda a vida adulta, conforme indicado pela reconstituição bem-sucedida do sistema imune em receptores adultos de transplante de medula óssea. É possível que o remanescente do timo involuído seja adequado para que ocorra alguma maturação das células T. Como as células T de memória possuem uma vida longa (talvez de mais de 20 anos nos seres humanos) e acumulam-se com o tempo, a necessidade de gerar novas células T diminui com a idade do indivíduo.
Os linfócitos T tem sua origem de precursores que se originam no fígado fetal e na medula óssea do adulto que migram para o timo. Esses precursores são progenitores multipotentes que entram no timo pela corrente sanguínea, cruzando o endotélio da vênula pós-capilar na região da junção corticomedular do timo. Nos camundongos, os linfócitos imaturos são inicialmente detectados no timo com 11 dias de uma gestação normal de 21 dias. Isso corresponde a cerca de 7 ou 8 semanas de gestação nos seres humanos. No timo, as células T em desenvolvimento são denominadas timócitos. Os timócitos mais imaturos são encontrados no seio subcapsular e na região cortical externa do timo. Desses locais, os timócitos migram para o córtex e através dele, onde ocorre a maioria dos eventos subsequentes de maturação. É no córtex que os timócitos expressam, pela primeira vez, os TCR γδ e αβ. As células T αβ amadurecem em células T CD4+ restritas pelo MHC da classe II ou em células T CD8+ restritas pelo MHC da classe I, à medida que deixam o córtex e entram na medula. Da medula, os timócitos positivos para CD4+ ou CD8+ deixam o timo por meio da circulação. Nas sessões seguintes, será discutida a maturação das células T αβ, enquanto as células T γδ são discutidas mais adiante, neste capítulo.
O ambiente do timo fornece estímulos que são necessários para a proliferação e a maturação dos timócitos. Muitos desses estímulos provêm de outras células tímicas distintas das células T em maturação. Dentro do córtex, as células epiteliais corticais do timo formam uma rede de longos processos citoplasmáticos, ao redor dos quais os timócitos precisam passar para alcançar a medula. Há também células epiteliais de um tipo distinto na medula, conhecidas como células epiteliais medulares do timo. As células dendríticas derivadas da medula óssea estão presentes na junção corticomedular e dentro da medula, enquanto os macrófagos são encontrados principalmente na medula. A migração dos timócitos por meio desse arranjo anatômico permite a ocorrência de interações físicas entre os timócitos e essas outras células, necessárias para a maturação e a seleção dos linfócitos T.
Dois tipos de moléculas produzidas pelas células do timo não linfoide são importantes para a maturação das células T. O primeiro tipo consiste em moléculas do MHC das classes I e II, expressas nas células epiteliais e células dendríticas no timo. As interações dos timócitos em desenvolvimento com essas moléculas do MHC dentro do timo são essenciais para a seleção do repertório de células T maduras, conforme discutido adiante. No segundo tipo, as células do estroma do timo, incluindo as células epiteliais, secretam citocinas e quimiocinas, que estimulam, respectivamente, a proliferação das células T imaturas e coordenam o trânsito dos timócitos de linhagem αβ em desenvolvimento do córtex para a medula. A mais bem definida dessas citocinas é a IL-7, mencionada anteriormente como um fator de crescimento linfopoético crítico. O movimento de células para dentro e através do timo é impulsionado por quimiocinas. Os progenitores dos linfócitos expressam o receptor de quimiocinas, CCR9, que se liga à quimiocina CCL25, que é produzida no córtex do timo. A entrada de precursores no timo depende da CCL25 e do CCR9. As quimiocinas, como a CCL21 e a CCL19, reconhecidas pelo receptor de quimiocinas CCR7 nos timócitos, medeiam o movimento direcionado das células T em desenvolvimento do córtex para a medula.
As taxas de proliferação celular e morte por apoptose são extremamente altas nos timócitos corticais. Um único precursor dá origem a uma grande progênie, e 95% dessas células morrem por apoptose antes de alcançar a medula. A morte celular deve-se a uma combinação de fatores, incluindo a incapacidade de efetuar um rearranjo produtivo do gene da cadeia β do TCR e, portanto, passar pelo ponto de controle de seleção de pré-TCR/β descrito adiante, a incapacidade de seleção positiva pelas moléculas do MHC no timo e a seleção negativa induzida por antígenos próprios (Figs. 8-3 e 8-4). Os timócitos corticais também são sensíveis à irradiação e aos glicocorticoides. In vivo, os glicocorticoides em altas doses induzem morte por apoptose dos timócitos corticais imaturos.
Estágios de Maturação das Células T
Durante a maturação das células T, existe uma ordem precisa no rearranjo dos genes dos TCR e na expressão dos correceptores CD4 e CD8 (Fig. 8-20 e também Fig. 8-19). No camundongo, a expressão do TCR γδ na superfície ocorre primeiro, dentro de 3 a 4 dias após a chegada das células precursoras no timo, enquanto o TCR αβ é expresso 2 ou 3 dias depois. No timo fetal humano, a expressão do TCR γδ começa em torno de 9 semanas de gestação, seguida da expressão do TCR αβ com 10 semanas.
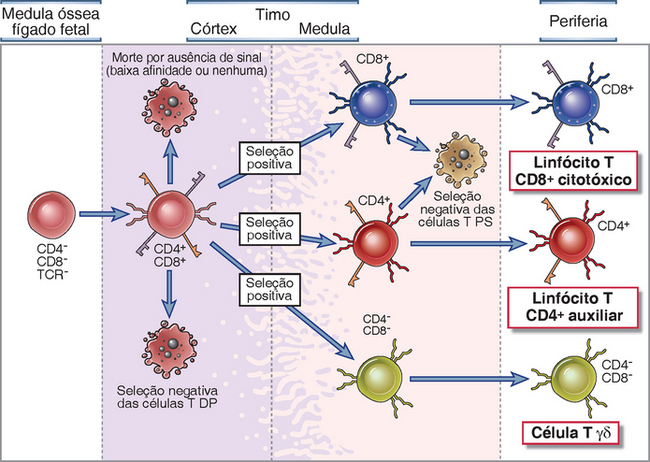
FIGURA 8-20 Maturação das células T no timo. Os precursores das células T saem da medula óssea e são transportados pela corrente sanguínea até o timo. No córtex do timo, os progenitores das células T αβ expressam TCR e correceptores CD4 e CD8. Os processos de seleção eliminam as células T autorreativas no córtex, no estágio duplo-positivo (DP), bem como os timócitos medulares positivos simples (PS). Promovem a sobrevida dos timócitos cujos TCR se ligam a moléculas próprias do MHC com baixa afinidade. A diferenciação funcional e fenotípica em células T CD4+CD8− ou CD8+CD4− ocorre na região medular, e as células T maduras são liberadas na circulação.
Timócitos Duplo-Negativos
Os timócitos corticais mais imaturos, que chegaram recentemente da medula óssea, contêm genes de TCR em sua configuração de linhagem germinativa e não expressam TCR, CD3, cadeias ζ, CD4 ou CD8; essas células são denominadas timócitos duplo-negativos. Nesse estágio, os timócitos também são considerados como no estágio de maturação de célula pró-T. A maioria (>90%) dos timócitos duplo-negativos que sobrevivem aos processos de seleção do timo dará origem, finalmente, a células T CD4+ e CD8+ que expressam TCR αβ, restritas pelo MHC, enquanto o restante dará origem a células T γδ. As proteínas Rag-1 e Rag-2 são expressas, pela primeira vez, no estágio de célula pró-T e são necessárias para o rearranjo dos genes do TCR. Os rearranjos Dβ-para-Jβ no lócus da cadeia β do TCR ocorrem primeiro; envolvem a junção do segmento gênico Dβ1 com um dos seis segmentos Jβ1, ou a junção do segmento Dβ2 com um dos seis segmentos Jβ2 (Fig. 8-21A). Os rearranjos Vβ-para-DJβ ocorrem na transição entre o estágio pró-T e o estágio subsequente pré-T durante o desenvolvimento da célula T αβ. As sequências de DNA entre os segmentos que sofrem rearranjo, incluindo os genes D, J e, possivelmente Cβ1 (se os segmentos Dβ2 e Jβ2 forem utilizados), sofrem deleção durante esse processo de rearranjo. Os produtos de transcrição nucleares primários dos genes β do TCR contêm o íntron entre o éxon VDJβ recombinado e o gene Cβ relevante. As caudas de poli-A são acrescentadas após a clivagem do transcrito primário de RNA em regiões suscetíveis à poliadenilação de consenso localizados em 3′ da região Cβ, enquanto as sequências entre o éxon VDJ e Cβ são emendadas para formar um mRNA maduro, em que os segmentos VDJ estão justapostos a um dos dois genes Cβ (dependendo do segmento J que foi selecionado durante o processo de rearranjo). A tradução desse mRNA dá origem a uma proteína da cadeia β do TCR integral. Os dois genes Cβ parecem ser intercambiáveis do ponto de vista funcional, e não há evidências de que uma célula T possa mudar de um gene C para outro. Além disso, o uso de qualquer um dos segmentos gênicos Cβ não influencia a função nem a especificidade do TCR. Os promotores nas regiões flanqueadoras 5′ dos genes Vβ atuam em conjunto com um amplificador potente, localizado em 3′ do gene Cβ2, após a aproximação dos genes V com o gene C por recombinação VDJ. Essa proximidade do promotor com o amplificador é responsável pela transcrição específica de alto nível do gene da cadeia β rearranjado do TCR na célula T.
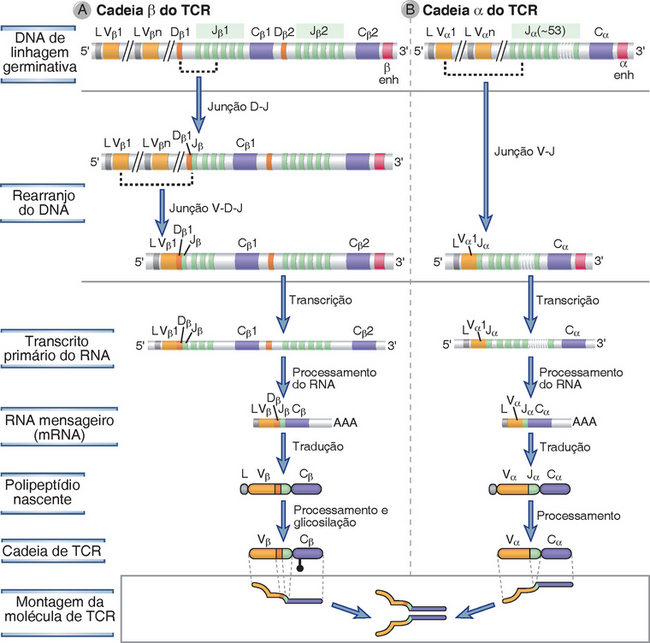
FIGURA 8-21 Recombinação e expressão dos genes das cadeias α e β do TCR. A sequência de recombinação e os eventos da expressão gênica são mostrados para a cadeia β do TCR (A) e para a cadeia α do TCR (B). No exemplo apresentado em A, a região variável (V) da cadeia β do TCR rearranjado inclui os segmentos gênicos Vβ1 e Dβ1 e o terceiro segmento J no agrupamento Jβ1. A região constante (C) é codificada pelo éxon Cβ1. Observe que, no lócus da cadeia β do TCR, o rearranjo começa com a junção D-para-J, seguida da junção V-para-DJ. Nos seres humanos, foram identificados 14 segmentos Jβ, porém nem todos são mostrados na figura. No exemplo mostrado em B, a região V da cadeia α do TCR inclui o gene Vα1 e o segundo segmento J no grupamento Jα (esse grupamento é composto de pelo menos 61 segmentos Jα nos seres humanos, porém nem todos são mostrados aqui).
Pré-receptor de Células T
Se ocorrer um rearranjo produtivo (i. e., síntese de uma cadeia polipeptídica com estrutura adequada) do gene da cadeia β do TCR em determinada célula pró-T, a proteína da cadeia β do TCR é expressa na superfície celular, em associação a uma proteína invariável, denominada pré-Tα, e às proteínas CD3 e ζ, para formar o receptor de células pré-T (pré-TCR) (Fig. 8-16B). O pré-TCR medeia a seleção das células pré-T em desenvolvimento, que efetuam um rearranjo produtivo da cadeia β do TCR. Cerca da metade de todas as células pré-T em desenvolvimento acrescenta ou remove bases nas junções do rearranjo, que são múltiplas de três (em pelo menos um cromossomo de β do TCR), e, portanto, aproximadamente metade de todas as células pré-T em desenvolvimento não expressa uma proteína β do TCR. A função do complexo pré-TCR no desenvolvimento da célula T é semelhante àquela do pré-BCR contendo cadeias leves substitutas no desenvolvimento das células B. Os sinais do pré-TCR medeiam a sobrevivência das células pré-T e contribuem para a maior expansão da proliferação durante o desenvolvimento das células T. Os sinais do pré-TCR também iniciam a recombinação no lócus da cadeia α do TCR e impulsionam a transição do estágio duplo-negativo de desenvolvimento do timócito para o estágio duplo-positivo (discutido adiante). Esses sinais também inibem o rearranjo adicional do lócus da cadeia β do TCR, limitando, em grande parte, a acessibilidade do outro alelo ao mecanismo de recombinação. Isso resulta na exclusão alélica da cadeia β (i. e., as células T maduras expressam apenas um dos dois alelos da cadeia β herdados). Como nas células pré-B, não se sabe qual ligante, se houver algum, é reconhecido pelo pré-TCR. Em geral, acredita-se que a sinalização do pré-TCR, como na sinalização do pré-BCR, seja iniciada de forma independente do ligante, dependendo do sucesso da montagem do complexo pré-TCR. A sinalização do pré-TCR é mediada por diversas cinases citosólicas e proteínas adaptadoras, também conhecidas pela sua ligação à sinalização do TCR (Cap. 7). A função essencial do pré-TCR na maturação das células T foi demonstrada por numerosos estudos realizados com camundongos, apresentando mutações genéticas, nas quais a falta de qualquer componente do complexo pré-TCR (i. e., a cadeia β do TCR, pré-Tα, CD3, ζ ou Lck) resulta em bloqueio da maturação das células T no estágio duplo-negativo.
Timócitos Duplo-positivos
No próximo estágio das células T, os timócitos expressam tanto CD4 como CD8 e são denominados timócitos duplo-positivos. A expressão de CD4 e CD8 é essencial para os eventos subsequentes de seleção, discutidos mais adiante. O rearranjo dos genes da cadeia α do TCR e a expressão dos heterodímeros αβ do TCR ocorrem na população de células duplo-positivas CD4+CD8+ pouco depois de a célula sobreviver ao ponto de controle do pré-TCR (Figs. 8-19 e 8-20). Uma segunda onda de expressão do gene Rag no final do estágio pré-T promove a recombinação do gene α do TCR. Como não há nenhum segmento D no lócus α do TCR, o rearranjo consiste apenas na junção dos segmentos V e J (Fig. 8-21B). O grande número de segmento Jα permite a realização de múltiplas tentativas na junção V-J produtiva em cada cromossomo, aumentando, assim, a probabilidade de que um TCR αβ funcional seja produzido. Ao contrário do lócus da cadeia β do TCR, no qual a produção de proteína e a formação do pré-TCR suprimem rearranjos adicionais, existe pouca ou nenhuma exclusão alélica no lócus de cadeia α. Assim, podem ocorrer rearranjos produtivos da cadeia α do TCR em ambos os cromossomos, e, se isso acontecer, a célula T irá expressar duas cadeias α. De fato, até 30% das células T maduras na periferia expressam dois TCR diferentes, com cadeias α diferentes, porém com a mesma cadeia β. É possível que apenas uma das duas cadeias α participe na formação de um complexo funcional entre TCR e o antígeno-específico. A regulação transcricional do gene da cadeia α ocorre de forma semelhante à da cadeia β. Existem promotores em 5′ de cada gene Vα, que possuem baixo nível de atividade e são responsáveis pela transcrição específica de alto nível da célula T quando colocados em proximidade a um amplificador da cadeia α, localizado em 3′ do gene Cα. A incapacidade de efetuar um rearranjo bem-sucedido da cadeia α do TCR, em qualquer um dos cromossomos, leva à falha da seleção positiva (discutida adiante). O timócito incapaz de efetuar um rearranjo produtivo do gene da cadeia α do TCR morre por apoptose.
A expressão do gene α do TCR no estágio duplo-positivo leva à formação do TCR αβ completo, que é expresso na superfície celular, em associação às proteínas CD3 e ζ. A expressão coordenada das proteínas CD3 e ζ e a montagem de complexos de TCR intactos são necessárias para a expressão de superfície. O rearranjo do gene α do TCR resulta em deleção do lócus δ do TCR situado entre os segmentos V (comum a ambos os loci α e δ) e os segmentos Jα (Fig. 8-7). Em consequência, essa célula T não é mais capaz de se transformar em uma célula T γδ e está totalmente comprometida com a linhagem de células T αβ. A expressão dos genes Rag e a recombinação adicional dos genes de TCR cessam após esse estágio de maturação.
As células duplo-positivas que sofrem processos de seleção bem-sucedidos amadurecem em células T CD4+ ou CD8+, que são denominadas timócitos simples-positivos. Assim, os estágios de maturação das células T no timo podem ser prontamente distinguidos pela expressão de CD4 e CD8 (Fig. 8-22A). Essa maturação fenotípica é acompanhada de maturação funcional. As células CD4+ adquirem a capacidade de produzir citocinas em resposta à estimulação antigênica subsequente e a expressar moléculas efetoras (como o ligante CD40), que “ajudam” os linfócitos B, as células dendríticas e os macrófagos, enquanto as células CD8+ tornam-se capazes de produzir moléculas que destroem outras células. Os timócitos simples-positivos maduros entram na medula do timo e, em seguida, deixam o timo para residir nos tecidos linfoides periféricos.
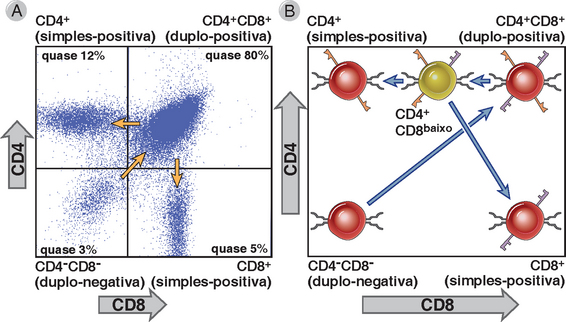
FIGURA 8-22 Expressão de CD4 e CD8 nos timócitos e seleção positiva nas células T do timo. A, A maturação dos timócitos pode ser seguida de alterações na expressão dos correceptores CD4 e CD8. A figura ilustra uma análise por citometria de fluxo de duas cores dos timócitos, utilizando anticorpos anti-CD4 e anti-CD8, cada um deles marcado com fluorocromo diferente. A porcentagem de todos os timócitos contribuindo para cada população é mostrada nos quatro quadrantes. O subgrupo menos maduro é constituído pelas células CD4−CD8−(duplo-negativas). As setas indicam a sequência de maturação. B, Seleção positiva das células T. As células T duplo-positivas diferenciam-se em um estágio CD4+CD8baixo e são instruídas a se transformarem em células CD4+ se o TCR em uma célula duplo-positiva reconhecer o MHC de classe II próprio com afinidade moderada e, portanto, receber sinais adequados do correceptor. Uma célula CD4+CD8baixo cujo TCR reconhece moléculas MHC da classe I falha ao receber sinais fortes do correceptor e diferencia-se em uma célula T CD8+, silenciando a expressão de CD4.
Processo de Seleção na Maturação das Células T αβ Restritas pelo MHC
A seleção de células T em desenvolvimento depende do reconhecimento do antígeno (complexos de peptídio – MHC) no timo e é responsável pela preservação das células úteis e eliminação das células potencialmente nocivas. O repertório de linfócitos T imaturos ou não selecionados consiste em células cujos receptores podem reconhecer qualquer antígeno peptídico (próprio ou estranho) apresentado por qualquer molécula do MHC (também própria ou estranha). Além disso, receptores podem teoricamente ser expressos, os quais não reconhecem nenhum complexo peptídio-molécula do MHC. Em todo o indivíduo, as únicas células úteis são as específicas para peptídeos estranhos apresentados pelas moléculas do MHC do indivíduo, isto é, moléculas próprias do MHC. Quando os timócitos duplo-positivos expressam pela primeira vez TCR αβ, esses receptores encontram peptídeos próprios (os únicos peptídeos normalmente presentes no timo) apresentados por moléculas do MHC do hospedeiro (as únicas moléculas do MHC disponíveis para apresentar peptídeos), principalmente nas células epiteliais do córtex do timo. O resultado desse reconhecimento é determinado principalmente pela força do encontro entre os TCR e os complexos de antígeno próprios-MHC. A seleção positiva é o processo que preserva as células T que reconhecem o MHC próprio (com peptídeos próprios) com baixa afinidade. Esse reconhecimento preserva as células que podem detectar antígenos apresentados pelas moléculas MHC desse indivíduo. Ao mesmo tempo, as células tornam-se comprometidas com a linhagem CD4 e CD8, com base no reconhecimento de moléculas do MHC da classe II ou da classe I, respectivamente, pelo TCR presente em cada célula. Além disso, em cada indivíduo, as células T que reconhecem os antígenos próprios com alta afinidade são potencialmente perigosas, visto que esse reconhecimento pode desencadear o processo de autoimunidade. A seleção negativa é o processo pelo qual os timócitos cujos TCR se ligam fortemente a antígenos peptídicos próprios em associação a moléculas do MHC próprias sofrem deleção (Fig. 8-20). O resultado final desses processos de seleção é que o repertório de células T maduras que deixam o timo é restrito pelo MHC próprio e tolerante a muitos antígenos próprios, enquanto apenas as células úteis completam o seu processo de maturação. Nas seções seguintes, serão discutidos os detalhes das seleções positiva e negativa.
Seleção Positiva dos Timócitos: Desenvolvimento do Repertório de Células T Restritas pelo MHC Próprio
A seleção positiva é o processo pelo qual os timócitos cujos TCR se ligam com baixa afinidade (i. e., fracamente) a complexos de peptídio próprio-MHC próprio são estimulados a sobreviver (Figs. 8-20 e 8-22). Os timócitos duplo-positivos são produzidos sem qualquer estimulação antigênica e começam a expressar TCR αβ com especificidades geradas de modo aleatório, o que faz com que algumas células T possuam TCR capazes de reconhecer estruturas próprias. No córtex do timo, essas células imaturas encontram células epiteliais, que apresentam uma variedade de peptídeos próprios ligados a moléculas do MHC da classe I e da classe II. O reconhecimento fraco desses complexos de peptídeo próprio-MHC próprio promove a sobrevida das células T. Os timócitos, cujos receptores não reconhecem moléculas do MHC próprio, morrem por uma via de omissão que leva à apoptose; esse fenômeno é denominado morte por negligência (Fig. 8-20). Assim, a seleção positiva assegura que as células T sejam restritas pelo MHC próprio.
Durante a transição das células duplo-positivas para positivas simples, os timócitos com TCR restritos pela classe I transformam-se em células CD8+CD4−, enquanto as células com TCR restritos pela classe II tornam-se CD4+CD8−. As células T imaturas duplo-positivas expressam TCR que podem reconhecer o MHC próprio da classe I ou da classe II. Foram propostos dois modelos para explicar o processo de comprometimento com a linhagem, cujo resultado consiste em coreceptores corretamente compatíveis com os TCR que reconhecem uma classe específica de moléculas do MHC. O modelo “estocástico” ou “probabilístico” sugere que o comprometimento das células T imaturas com uma das linhagens depende da probabilidade aleatória de diferenciação de uma célula duplo-positiva em uma célula T CD4+ ou CD8+. Nesse modelo, uma célula que reconhece o MHC próprio da classe I pode se diferenciar aleatoriamente em uma célula T CD8+ (com o coreceptor apropriado) e sobreviver, ou pode se diferenciar aleatoriamente em uma célula T CD4+ (com o correceptor “incorreto”), que pode não receber sinais de sobrevida. Nesse processo de diferenciação aleatória em células positivas simples, o coreceptor não é compatível com o reconhecimento da classe correta de moléculas do MHC em cerca de metade das vezes. Uma hipótese mais amplamente aceita é a de que o processo de comprometimento com a linhagem ligada à seleção positiva não é aleatório, porém “instrucional”. Os modelos instrucionais sugerem que os TCR restritos pela classe I e pela classe II emitem sinais diferentes, que induzem ativamente a expressão do coreceptor correto, enquanto eliminam a expressão do outro coreceptor. Sabe-se que as células duplo-positivas passam por um estágio em que expressam altos níveis de CD4 e baixos níveis de CD8. Se o TCR presente nessa célula for restrito pelo MHC da classe I, quando ele identificar o MHC da classe I apropriado e o peptídio próprio, receberá um sinal fraco, visto que os níveis do coreceptor CD8 estão baixos, e, além disso, a molécula de CD8 associa-se bem menos à tirosinocinase Lck do que as CD4. Esses sinais fracos ativam os fatores de transcrição (como o Runx3), que estimulam a expressão de CD8, gerando uma célula T CD8+ restrita pela classe I. Por sua vez, se o TCR na célula for restrito pela classe II, quando ele identificar a classe II, receberá um sinal mais potente, visto que os níveis de CD4 estão elevados, e a molécula de CD4 associa-se relativamente bem a Lck. Esses sinais fortes ativam um conjunto diferente de fatores de transcrição (incluindo ThPok), que estimulam a expressão de CD4 e eliminam CD8.
Os peptídeos ligados a moléculas do MHC nas células epiteliais do timo desempenham um papel essencial na seleção positiva. No Capítulo 6, será descrito como as moléculas de superfície celular do MHC da classe I e da classe II sempre contêm peptídeos ligados. Esses peptídeos associados ao MHC nas células apresentadoras de antígenos no timo provavelmente desempenham dois papéis na seleção positiva – em primeiro lugar, promovem a expressão estável das moléculas do MHC na superfície celular, e, em segundo lugar, podem influenciar as especificidades das células T selecionadas. É também evidente, de uma variedade de estudos experimentais, que alguns peptídeos são melhores do que outros para sustentar a seleção positiva, e diferentes peptídeos diferem nos repertórios de células T que eles selecionam. Esses resultados sugerem que o reconhecimento de um antígeno específico, e não apenas o reconhecimento de MHC, desempenha algum papel na seleção positiva. Uma consequência da seleção positiva induzida por peptídeos próprios é que as células que amadurecem têm a capacidade de reconhecer os peptídeos próprios. No Capítulo 2, abordou-se que a sobrevida dos linfócitos virgens antes de seu encontro com antígenos estranhos requer sinais de sobrevivência, aparentemente gerados pelo reconhecimento de antígenos próprios nos órgãos linfoides periféricos. Os mesmos peptídeos próprios que medeiam a seleção positiva dos timócitos duplo-positivos no timo podem estar envolvidos na manutenção das células T maduras (positivas simples) virgens vivas nos órgãos periféricos, como os gânglios linfáticos e o baço.
O modelo de seleção positiva com base no reconhecimento fraco de antígenos próprios levanta uma questão fundamental: como a seleção positiva, impulsionada por antígenos próprios, produz um repertório de células T maduras específicas para antígenos estranhos? A resposta provável é que a seleção positiva permite a sobrevida e a diferenciação de muitos clones diferentes de células T, e muitas dessas células T, que reconhecem peptídeos próprios com baixa afinidade, irão, após a sua maturação, reconhecer de modo casual peptídeos estranhos com afinidade alta o suficiente para serem ativados e gerar respostas imunológicas.
Seleção Negativa dos Timócitos: Tolerância Central
Os timócitos cujos receptores reconhecem, com alta afinidade, complexos de peptídio-MHC no timo sofrem apoptose (denominada seleção negativa) ou diferenciam-se em células T reguladoras (Fig. 8-20). Entre as células T duplo-positivas geradas no timo, algumas podem expressar TCR que reconhecem antígenos próprios com alta afinidade. Os peptídeos presentes no timo são autopeptídios, derivados de antígenos proteicos amplamente expressos, bem como de algumas proteínas que, acredita-se, sejam restritas a determinados tecidos. (Lembre-se de que os micro-organismos que entram através das vias comuns, isto é, os epitélios, são capturados e transportados até os gânglios linfáticos e não tendem a entrar no timo.) Nas células T imaturas, uma importante consequência do reconhecimento do antígeno com alta afinidade é a deflagração do processo de apoptose, que leva à morte ou à deleção das células. Assim, muitos dos timócitos imaturos que expressam receptores de alta afinidade para antígenos próprios no timo são eliminados, resultando na seleção negativa do repertório de células T. Esse processo elimina as células T potencialmente autorreativas mais nocivas e constitui um dos mecanismos que assegura que o sistema imune não irá responder a muitos antígenos próprios, denominado autotolerância. A tolerância induzida nos linfócitos imaturos pelo reconhecimento de antígenos próprios nos órgãos linfoides geradores (ou centrais) é também denominada tolerância central, para contrastá-la com a tolerância periférica induzida nos linfócitos maduros por antígenos próprios nos tecidos periféricos. No Capítulo 14, serão discutidos com mais detalhes os mecanismos e a importância fisiológica da intolerância imunológica.
A deleção das células T imaturas autorreativas pode ocorrer tanto no estágio duplo-positivo no córtex como em células T simples-positivas recém-geradas na medula. As células apresentadoras de antígenos no timo que medeiam a seleção negativa consistem principalmente em células dendríticas e macrófagos derivados da medula óssea, ambos abundantes na medula, e em células epiteliais da medula do timo, enquanto as células epiteliais corticais são especialmente (e talvez,únicas) efetivas na indução da seleção positiva. As células T duplo-positivas são atraídas até a medula do timo pelas quimiocinas específicas do CCR7, a CCL21 e a CCL19. Na medula, as células epiteliais medulares do timo expressam uma proteína nuclear, denominada AIRE (regulador autoimune), que induz a expressão de diversos genes específicos de diferentes tecidos no timo. Os produtos desses genes são expressos normalmente apenas em órgãos periféricos específicos, como o pâncreas e a tireoide. A expressão dessas moléculas dependentes do AIRE no timo permite que sejam disponibilizados muitos peptídeos específicos de tecidos para a sua apresentação às células T em desenvolvimento, facilitando a deleção (seleção negativa) dessas células. Uma mutação no gene que codifica o AIRE resulta em uma síndrome poliendócrina autoimune, ressaltando a importância do AIRE em mediar a tolerância central a antígenos específicos de tecidos (Cap. 14).
O mecanismo da seleção negativa no timo é a indução da morte por apoptose. Ao contrário do fenômeno de morte por negligência que ocorre na ausência de seleção positiva, na seleção negativa, são gerados sinais ativos que promovem a morte quando o TCR dos timócitos imaturos liga-se com alta afinidade a um antígeno. A indução de uma proteína pró-apoptótica, denominada Bim, pela sinalização do TCR provavelmente desempenha um papel crucial na indução da permeabilidade mitocondrial e na apoptose dos timócitos durante a seleção negativa (Cap. 14). É também evidente que o reconhecimento de um antígeno com alta afinidade pelas células T imaturas deflagra o processo de apoptose; entretanto, o reconhecimento desses mesmos peptídeos pelos linfócitos maduros (em associação a outros sinais, Cap. 9) inicia respostas das células T. A base bioquímica dessa diferença fundamental ainda não foi definida.
Reconhecimento de antígenos próprios no timo pode gerar uma população de células T reguladoras, cuja função é impedir a ocorrência de reações autoimunes (Cap. 14). Não foi esclarecido quais fatores determinam a escolha entre os dois destinos alternativos das células T imaturas que reconhecem antígenos próprios com alta afinidade, isto é, a deleção das células T imaturas e o desenvolvimento das células T reguladoras. É possível que interações de afinidade ligeiramente menores do que as necessárias para a deleção possam levar ao desenvolvimento das células T reguladoras naturais, porém é necessário obter evidências claras para essa hipótese.
Linfócitos T γδ
Os timócitos que expressam TCR αβ e γδ consistem em linhagens separadas com um precursor comum. No timo fetal, os primeiros rearranjos dos genes dos TCR envolvem os loci γ e δ. A recombinação dos loci γ e δ dos TCR ocorre de modo semelhante a outros rearranjos dos genes dos receptores de antígenos, embora a ordem do rearranjo pareça ser menos rígida do que em outros loci. Na célula T duplo-negativa em desenvolvimento, o rearranjo dos loci β, γ ou δ dos TCR é inicialmente possível. Se uma célula tem sucesso no rearranjo produtivo do lócus γ do TCR, bem como dos loci δ, antes de realizar um rearranjo produtivo de β do TCR, ela é selecionada na linhagem de células T γδ. Isso ocorre em cerca de 10% das células T duplo-negativas em desenvolvimento. Em cerca de 90% dos casos, ocorre inicialmente um rearranjo produtivo do gene β do TCR. Nessa situação, a sinalização do pré-TCR seleciona essas células para se transformarem na linhagem de células T αβ, e a deleção final do δ do TCR, quando o gene α do TCR estiver rearranjado (o lócus do gene δ do TCR está inserido no lócus do gene α do TCR) resulta em comprometimento irreversível da linhagem αβ.
A diversidade do repertório das células T γδ é teoricamente ainda maior do que a das células T αβ, em parte pelo fato de que as sequências de reconhecimento de heptâmero-nonâmero adjacentes aos segmentos D permitindo a junção D-para-D. Entretanto, paradoxalmente, a diversidade real dos TCR γδ expressos é limitada, visto que apenas alguns dos segmentos V, D e J disponíveis são utilizados nas células T γδ maduras, por razões desconhecidas. Essa diversidade limitada lembra a diversidade limitada do subgrupo B-1 de linfócitos B e está de acordo com o conceito de que as células T γδ servem de defesa inicial contra um número limitado de micro-organismos comumente encontrados nas barreiras epiteliais.
Células NKT
As células NKT não são restritas ao MHC e não reconhecem peptídeos apresentados pelas células apresentadoras de antígenos. Essas células expressam TCR αβ que são restritos por CD1 e também exibem um marcador de superfície encontrado nas células NK, explicando o seu nome. Os TCR das células NKT reconhecem antígenos lipídicos apresentados por moléculas CD1. As moléculas CD1 são semelhantes ao MHC da classe I, compostas de uma cadeia pesada e de β2 microglobulina. A cadeia pesada possui uma fenda, onde se encaixa o ligante antigênico, constituído de resíduos hidrofóbicos, que pode se ligar a antígenos lipídicos e apresentá-los. Esses antígenos lipídicos podem derivar de micro-organismos que sofreram endocitose ou podem consistir em lipídios próprios (Cap. 6). No córtex do timo, as células T αβ duplo-positivas que expressam receptores de células T que reconhecem moléculas CD1 expressas em timócitos duplo-positivos adjacentes são induzidas a se diferenciar em células NKT. Um grande número de células NKT restritas por CD1 apresenta um TCR “invariável”, resultante de um rearranjo singular e estereotípico do gene da cadeia α do TCR. As células NKT secretam citocinas e participam na defesa do hospedeiro e também podem atuar para regular uma variedade de respostas imunológicas. As funções das células NKT são descritas no Capítulo 10.
• Os linfócitos B e T originam-se de um precursor comum derivado da medula óssea, que se compromete com a linhagem linfocitária. A maturação das células B prossegue na medula óssea, enquanto os progenitores iniciais das células T migram para o timo, onde completam a sua maturação. A maturação inicial caracteriza-se por proliferação celular induzida por citocinas, principalmente a IL-7, levando a uma expansão do número de linfócitos que acabaram de se comprometer com linhagens individuais.
• Os sinais extracelulares induzem a ativação de fatores de transcrição, que estimulam a expressão de genes específicos de linhagem e abrem loci gênicos de receptores de antígenos específicos no nível de acessibilidade da cromatina.
• O desenvolvimento das células B e T envolve o rearranjo somático de segmentos gênicos dos receptores de antígenos e a expressão inicial da proteína μ da cadeia pesada das Ig nos precursores das células B e moléculas β do TCR nos precursores das células T. A expressão inicial dos pré-receptores de antígenos e a expressão subsequente dos receptores de antígenos são essenciais para a sobrevida, a expansão e a maturação dos linfócitos em desenvolvimento, bem como para os processos de seleção que levam a um repertório diversidade de especificidades antigênicas úteis.
• Os receptores de antígenos das células B e T são codificados por genes de receptores, formados por um número limitado de segmentos gênicos, espacialmente segregados nos loci dos receptores de antígenos da linhagem germinativa, mas somaticamente recombinados nas células B e T em desenvolvimento.
• Os loci separados codificam a cadeia pesada, a cadeia leve κ e a cadeia leve λ das Ig, bem como a cadeia β, as cadeias α e δ e a cadeia γ dos TCRs. Esses loci contêm segmentos V, J e, apenas nos loci da cadeia pesada das Ig e β e δ dos TCR, são encontrados segmentos gênicos D. Os segmentos J localizam-se imediatamente adjacentes aos éxons que codificam os domínios constantes, enquanto os segmentos V situam-se a uma grande distância dos segmentos J. Quando presentes, os segmentos D estão localizados entre os grupamentos VJ. O rearranjo somático dos loci das Ig e dos TCRs envolve a junção dos segmentos D e J nos loci que contêm segmentos D, seguido da junção do segmento V com os segmentos DJ recombinados nesses loci, ou de junção V-para-J direta nos outros loci.
• Esse processo de recombinação gênica somática é mediado por um complexo enzimático recombinase, que inclui os componentes específicos dos linfócitos, as proteínas Rag-1 e Rag-2.
• A diversidade dos repertórios de anticorpos e de TCR é gerada pelas associações combinatórias de múltiplos genes V, D e J da linhagem germinativa, enquanto a diversidade juncional é gerada pela adição ou remoção aleatórias de nucleotídios nos locais de recombinação. Esses mecanismos geram a maior diversidade nas junções dos segmentos que formam a terceira região hipervariável dos polipeptídios de anticorpos e de TCR.
• A maturação das células B ocorre em estágios, que se caracterizam por diferentes padrões de rearranjo e expressão dos genes das Ig. Nos precursores iniciais das células B, denominados células pró-B, os genes das Ig estão inicialmente na configuração da linhagem germinativa, e ocorre rearranjo D para J no lócus das cadeias pesadas de Ig.
• Na transição da célula pró-B para a célula pré-B, a recombinação V-D-J é completada no lócus da cadeia H das Ig. Ocorre produção de um transcrito primária de RNA contendo o éxon VDJ e os éxons do gene C das Ig, e o éxon VDJ é emendado aos éxons da região C μ do RNA da cadeia pesada, para gerar um mRNA maduro que é traduzido na proteína da cadeia pesada μ. O pré-BCR é formado pelo emparelhamento da cadeia μ com cadeias leves substitutas e pela associação às moléculas de sinalização Igα e Igβ. Esse receptor emite sinais de sobrevivência e de proliferação, bem como sinais para inibir o rearranjo do outro alelo da cadeia pesada (exclusão alélica).
• À medida que as células se diferenciam em células B imaturas, ocorre recombinação V-J inicialmente no lócus κ das Ig, e há expressão das proteínas da cadeia leve. Em seguida, ocorre montagem das cadeias pesadas e leves para formar moléculas intactas de IgM, expressas na superfície celular. As células B imaturas deixam a medula óssea para ocupar tecidos linfoides periféricos, onde elas completam o seu processo de maturação. No estágio de célula B madura, a síntese das cadeias pesadas μ e δ ocorre em paralelo, mediada através do processamento (splicing) alternativo dos produtos de transcrição primária do RNA das cadeias pesadas, permitindo assim a expressão tanto da IgM quanto da IgD na membrana.
• Durante a maturação dos linfócitos B, as células B imaturas que expressam receptores de antígenos com alta afinidade, específicos para antígenos próprios presentes na medula óssea, são induzidas a editar seus genes de receptores ou são eliminadas. A edição do receptor pode envolver um rearranjo adicional no lócus κ das Ig e, por fim, envolver também o rearranjo do gene da cadeia leve λ das Ig. As células B que expressam cadeias leves λ são, com frequência, células que sofreram edição do receptor.
• A maturação das células T no timo também progride em estágios, que se distinguem pelo padrão de expressão dos genes de receptores de antígenos, pelas moléculas dos correceptores CD4 e CD8 e pela sua localização no timo. Os primeiros imigrantes da linhagem T para o timo não expressam TCR nem moléculas CD4 ou CD8. As células T em desenvolvimento no timo, denominadas timócitos, ocupam inicialmente a porção externa do córtex, onde sofrem proliferação, rearranjo dos genes dos TCR e expressão das moléculas CD3, TCR, CD4 e CD8 na superfície. À medida que as células amadurecem, migram do córtex para a medula.
• Os timócitos mais imaturos, denominados células pró-T, são CD4− CD8− (duplo-negativas), e os genes do TCR estão inicialmente na configuração da linhagem germinativa nesse estágio. Nesse estágio, ocorre rearranjo dos genes das cadeias β, δ e γ dos TCR.
• No estágio de célula pré-T, os timócitos permanecem duplo-negativos, porém a recombinação V-D-J é completada no lócus da cadeia β do TCR. Os produtos de transcrição primários da cadeia β são expressos e processados para aproximar um éxon VDJ adjacente a um segmento Cβ, e os polipeptídios da cadeia β do TCR são produzidos. A cadeia β do TCR associa-se à proteína pré-Tα invariável para formar um pré-TCR. O pré-TCR transduz sinais que inibem o rearranjo do outro alelo da cadeia β (exclusão alélica) e promovem a diferenciação para o estágio de dupla expressão de CD4 e CD8 e a proliferação adicional dos timócitos imaturos. No estágio CD4+CD8+ (duplo-positivo) de desenvolvimento da célula T, ocorre recombinação V-J no lócus da cadeia α do TCR, são produzidos os polipeptídios da cadeia α e baixos níveis do TCR são expressos na superfície celular.
• Os processos de seleção impulsionam a maturação dos timócitos duplo-positivos que expressam TCR e formatam o repertório de células T para restrição pelo MHC próprio e autotolerância.
• A seleção positiva dos timócitos CD4+CD8+ TCR αβ exige o reconhecimento de complexos de peptídio-MHC com baixa afinidade nas células epiteliais do timo, levando a um resgate das células da morte programada. À medida que os timócitos TCR αβ amadurecem, eles migram para a medula e transformam-se em células CD4+CD8− ou CD8+CD4−. A seleção positiva é acompanhada de comprometimento com a linhagem. Isso resulta na compatibilidade dos TCR, que reconhecem o MHC da classe I com expressão de CD8 e silenciamento de CD4; os TCR que reconhecem moléculas do MHC da classe II são compatíveis com a expressão de CD4 e a perda da expressão de CD8.
• A seleção negativa dos timócitos duplo-positivos CD4+ CD8+ TCR αβ ocorre quando essas células reconhecem com alta afinidade antígenos presentes no timo. Esse processo é responsável pela tolerância a muitos antígenos próprios. Os timócitos da medula continuam sendo selecionados de modo negativo, e as células que não sofrem deleção clonal adquirem a capacidade de se diferenciar em células T CD4+ ou CD8+ virgens e, finalmente, migram para os tecidos linfoides periféricos.
Desenvolvimento Inicial das Células B e Recombinação V(D)J
Cobaleda C, Busslinger M. Developmental plasticity of lym- phocytes. Current Opinion in Immunology. 2008;20:139-148.
Jenkinson EJ, Jenkinson WE, Rossi SW, Anderson G. The thymus and T-cell commitment: the right choice for Notch? Nature Reviews Immunology. 2006;6:551-555.
Johnson K, Reddy KL, Singh H. Molecular pathways and mechanisms regulating the recombination of immunoglobulin genes during B-lymphocyte development. Advances in Experimental Medicine and Biology. 2009;650:133-147.
Jung D, Giallourakis C, Mostoslavsky R, Alt FW. Mechanism and control of V(D)J recombination at the immunoglobulin heavy chain locus. Annual Review of Immunology. 2006;24:541-570.
Nemazee D. Receptor editing in lymphocyte development and central tolerance. Nature Reviews Immunology. 2006;6:728-740.
Perlot T, Alt FW. Cis-regulatory elements and epigenetic changes control genomic rearrangements of the IgH locus. Advances in Immunology. 2008;99:1-32.
Schatz DG, Y JI. Recombination centers and the orchestration of V(D)J recombination. Nature Reviews Immunology. 11, March 2011.
Boehm T. Thymus development and function. Current Opinion in Immunology. 2008;20:178-184.
Carpenter AC, Bosselut R. Decision checkpoints in the thymus. Nature Immunology. 2010;11:666-673.
Godfrey DI, Stankovic S, Baxter AG. Raising the NKT cell family. Nature Immunology. 2010;11:197-206.
He X, Park K, Kappes DJ. The role of ThPOK in control of CD4/CD8 lineage commitment. Annual Review of Immunology. 2010;28:295-320.
Kyewski B, Klein L. A central role for central tolerance. Annual Review of Immunology. 2006;24:571-606.
Maillard I, Fang T, Pear WS. Regulation of lymphoid development, differentiation, and function by the Notch pathway. Annual Review of Immunology. 2005;23:945-974.
Rodewald HR. Thymus organogenesis. Annual Review of Immunology. 2008;26:355-388.
Rothenberg EV, Moore JE, Yui MA. Launching the T-cell-lineage developmental programme. Nature Reviews Immunology. 2008;8:9-21.
Singer A, Adoro S, Park JH. Lineage fate and intense debate: myths, models and mechanisms of CD4 versus CD8 lineage choice. Nature Reviews Immunology. 2008;8:788-801.
von Boehmer H, Aifantis I, Gounari F, Azugui O, Haughn L, Apostolou I, Jaeckel E, Grassi F, Klein L. Thymic selection revisited: how essential is it? Immunological Reviews. 2003;191:62-78.
MicroRNA e Desenvolvimento dos Linfócitos
Hoefig KP, Heissmeyer V. MicroRNAs grow up in the immune system. Current Opinion in Immunology. 2008;20:281-287.
Lodish HF, Zhou B, Liu G, Chen CZ. Micromanagement of the immune system by microRNAs. Nature Reviews Immunology. 2008;8:120-130.
Xiao C, Rajewsky K. MicroRNA control in the immune system: basic principles. Cell. 2009;136:26-36.