APÊNDICE IV TÉCNICAS DE LABORATÓRIO COMUMENTE USADAS EM IMUNOLOGIA
MÉTODOS DE LABORATÓRIO USANDO ANTICORPOS,
CAMUNDONGOS TRANSGÊNICOS E ALVOS GENÉTICOS,
MÉTODOS PARA O ESTUDO DE RESPOSTAS DE LINFÓCITOS T,
MÉTODOS PARA O ESTUDO DE RESPOSTAS DE LINFÓCITOS B,
Muitas técnicas laboratoriais, que são rotineiras em ambientes clínicos e de pesquisa, são baseadas no uso de anticorpos. Além disso, muitas das técnicas da biologia molecular moderna forneceram informações valiosas sobre o sistema imunológico. Mencionamos estas técnicas com frequência neste livro. Neste apêndice, descrevemos os princípios subjacentes aos métodos laboratoriais mais comumente usados em imunologia. Além disso, resumimos como respostas de linfócitos T e B são estudadas com o uso de técnicas laboratoriais. Detalhes sobre como conduzir diversos ensaios podem ser encontrados em manuais de laboratório.
MÉTODOS DE LABORATÓRIO USANDO ANTICORPOS
A especificidade primorosa dos anticorpos para antígenos específicos transforma os anticorpos em valiosos reagentes para a detecção, purificação e quantificação de antígenos. Como anticorpos podem ser produzidos contra quase qualquer tipo de macromolécula e pequenas estruturas químicas, técnicas baseadas em anticorpos podem ser usadas para estudar praticamente qualquer tipo de molécula em solução ou em células. O método para produzir anticorpos monoclonais (Cap. 5) aumentou em muito nossa habilidade de produzir anticorpos para praticamente qualquer especificidade desejada. Historicamente, muitos dos usos de anticorpos dependiam da habilidade do anticorpo e do antígeno específico de formar grandes complexos imunológicos, em soluções ou em géis, que podiam ser detectados por diversos métodos ópticos. Estes métodos tiveram grande importância nos estudos iniciais, mas foram quase totalmente substituídos por métodos mais simples baseados em anticorpos ou antígenos imobilizados.
Quantificação de Antígenos por Imunoensaios
Métodos imunológicos de quantificação da concentração de antígeno oferecem sensibilidade e especificidade formidáveis e se tornaram técnicas padrão tanto para pesquisas como para aplicações clínicas. Todos os métodos modernos de quantificação imunoquímica são baseados em ter um antígeno ou anticorpo puro cuja quantidade pode ser medida por uma molécula indicadora (ou um marcador). Quando o antígeno ou anticorpo é marcado com um radioisótopo, conforme introduzido por Rosalyn Yalow e colegas, ele pode ser quantificado por instrumentos que detectam eventos de decomposição radioativa; o ensaio é chamado de ensaio radioimunológico (RIA). Quando o antígeno ou anticorpo é ligado covalentemente a uma enzima, ele pode ser quantificado por determinação espectrofotométrica da taxa de conversão na qual a enzima converte um substrato cristalino em um produto colorido; o ensaio é chamado de ensaio imunoabsorvente ligado à enzima (ELISA). Existem diversas variações de RIA e ELISA, mas a versão do ensaio usada mais comumente é o ensaio sanduíche (Fig. A-1). O ensaio sanduíche usa dois anticorpos reativos diferentes com epitopos diferentes no antígeno cuja concentração deve ser determinada. Uma quantidade fixa de um anticorpo é ligada a uma série de suportes sólidos replicados, como tiras plásticas de microtitulação. Soluções de amostra contendo antígeno a uma concentração desconhecida ou uma série de soluções padrão com concentrações de antígeno conhecidas são acrescentadas às tiras e deixadas para que a ligação ocorra. Antígeno não ligado é removido por lavagem, e o segundo anticorpo, que é ligado à enzima ou radio-marcado, é adicionado para se ligar. O antígeno serve como uma ponte, então quanto mais antígeno nas soluções padrão ou de teste, mais segundos anticorpos ligados à enzima ou radiomarcados irão se ligar. Os resultados das soluções padrão são usados para construir uma curva de ligação para o segundo anticorpo como função da concentração de antígeno, da qual podem ser inferidas as quantidades de antígeno nas soluções de amostra. Quando este teste é realizado com dois anticorpos monoclonais, é essencial que estes anticorpos vejam os determinantes não sobrepostos no antígeno; de outra maneira, o segundo anticorpo não pode se ligar.
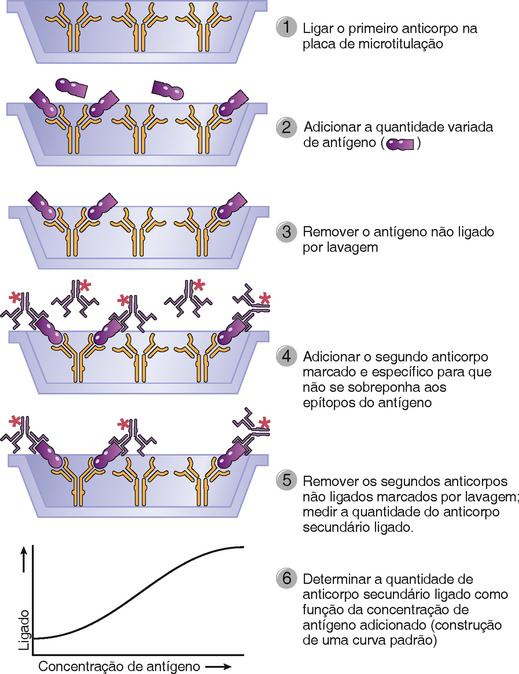
FIGURA A-1 Ensaio sanduíche imunoabsorvente ligado à enzima ou radioimunoensaio. Uma quantidade fixa de um anticorpo imobilizado é usada para capturar um antígeno. A ligação de um segundo anticorpo marcado que reconhece um determinante não sobreposto no antígeno aumentará conforme a concentração do antígeno aumenta, permitindo assim a quantificação do antígeno.
Em uma variante clínica importante de ensaios de imunoligação, amostras de pacientes podem ser testadas para a presença de anticorpos que são específicos para um antígeno microbiano (p. ex., anticorpos reativos com proteínas do vírus da imunodeficiência humana [HIV] ou o vírus da hepatite B) como indicadores de infecção. Neste caso, uma quantidade saturante de antígeno é adicionada para replicar em poços contendo anticorpos ligados à placa ou o antígeno fica diretamente ligado à placa, e então diluições em série do soro do paciente são adicionados para se ligar. A quantidade de anticorpos do paciente ligada ao antígeno imobilizado é determinada pelo uso de um segundo anticorpo anti-imunoglobulina (Ig) humano ligado à enzima ou radiomarcado.
Identificação e Purificação de Proteínas
Anticorpos podem ser usados para identificar e caracterizar proteínas e purificar proteínas específicas de misturas. Dois métodos comumente usados para identificar e purificar proteínas são a imunoprecipitação e a cromatografia de imunoafinidade. O Western Blotting é uma técnica imunológica para determinar a presença de uma proteína em uma amostra biológica.
Imunoprecipitação e Cromatografia de Imunoafinidade
A imunoprecipitação é uma técnica na qual um anticorpo específico para um antígeno proteico em uma mistura de proteínas é usado para identificar este antígeno específico (Fig. A-2A). O anticorpo é normalmente adicionado a uma mistura de proteínas (geralmente um lisado por detergente de células específicas), e proteína estafilocócica A (ou proteína G) ligadas covalentemente a contas de agarose é acrescentada à mistura. As porções Fab do anticorpo se ligam à proteína-alvo, e a porção Fc do anticorpo é capturada pela proteína A ou proteína G nas contas. Proteínas indesejadas que não se ligam ao anticorpo são então removidas lavando as contas (por adição de detergente e centrifugação repetidas). A proteína específica que é reconhecida e está agora ligada ao anticorpo pode ser eluída das contas e dissociada do anticorpo com o uso de um desnaturante (como o dodecil sulfato de sódio), e as proteínas são separadas por eletroforese em gel de poliacrilamida-dodecil sulfato de sódio (SDS-PAGE). Proteínas podem ser detectadas após a electroforese tingindo o gel de poliacrilamida com uma coloração de proteína ou por análise de Western Blotting (descrita mais adiante). Se a mistura original contivesse proteínas marcadas radioativamente, as proteínas específicas imunoprecipitadas pelo anticorpo poderiam ser reveladas por autofluorografia ou autoradiografia, onde as bandas de proteína são capturadas em filme de raios X colocado sobre o gel poliacrilamida-SDS seco contendo proteínas separadas.
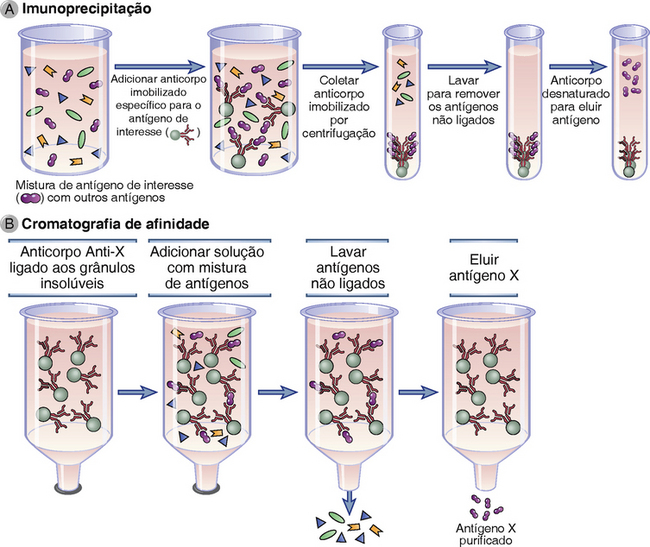
FIGURA A-2 Isolamento de um antígeno por imunoprecipitação ou cromatografia de afinidade. A, Um antígeno específico pode ser purificado de uma mistura de antígenos em soro ou em outras soluções pela adição de anticorpos específicos ao antígeno que são ligados a contas insolúveis. Antígenos livres são lavados, e o antígeno desejado é recuperado pela alteração do pH ou da força iônica da solução de modo que a afinidade da ligação anticorpo-antígeno é diminuída. A imunoprecipitação pode ser usada como um meio de purificação, como um meio de quantificação, ou como um meio de identificação de um antígeno. Antígenos purificados por imunoprecipitação são frequentemente analisados por eletroforese de gel de poliacrilamida-dodecil sulfato de sódio. B, A cromatografia de afinidade tem como base o mesmo princípio da imunoprecipitação, exceto que o anticorpo é fixado a uma matriz ou contas insolúveis, geralmente em uma coluna. O método é usado com frequência para isolar antígenos solúveis (mostrado) ou anticorpos específicos para um antígeno imobilizado.
A cromatografia de imunoafinidade, uma variante da cromatografia de afinidade, é um método de purificação que depende de anticorpos ligados a um suporte insolúvel para purificar antígenos de uma solução (Fig. A-2B). Anticorpos específicos para o antígeno desejado são normalmente ligados covalentemente a um suporte sólido, como contas de agarose, e dispostos em uma coluna. Uma mistura complexa de antígenos é passada pelas contas permitindo que o antígeno reconhecido pelo anticorpo se ligue. Moléculas não ligadas são lavadas, e o antígeno ligado é eluído por alteração do pH ou por exposição à alta salinidade ou outras condições caotrópicas que rompem as interações antígeno-anticorpo. Um método semelhante pode ser usado para purificar anticorpos de sobrenadantes da cultura ou fluidos naturais, como soro, ligando primeiro o antígeno às contas e passando os sobrenadantes ou soro.
Western Blotting
Western Blotting (Fig. A-3) é usado para identificar e determinar a quantidade relativa e o peso molecular de uma proteína em uma mistura de proteínas ou de outras moléculas. Primeiro a mistura é sujeita à separação analítica, normalmente por SDS-PAGE, de modo que as posições finais de proteínas diferentes no gel são uma função do seu tamanho molecular. A coleção de proteínas é então transferida, por eletroforese, do gel separador de poliacrilamida para uma membrana de suporte de forma que a membrana adquire uma réplica da coleção de macromoléculas separadas presentes no gel. SDS é deslocado da proteína durante o processo de transferência, e frequentemente determinantes antigênicos nativos são recuperados conforme a proteína se redobra. A posição do antígeno proteico na membrana pode então ser detectada pela ligação de um anticorpo específico não marcado para aquela proteína (o anticorpo primário) seguido por um segundo anticorpo marcado que se liga ao anticorpo primário. Esta abordagem fornece informações sobre o tamanho e a quantidade de antígenos. No geral, sondas do segundo anticorpo são marcadas com enzimas que geram sinais quimiluminescentes e deixam imagens em filme fotográfico. Fluoróforos próximos do infravermelho também podem ser usados para marcar anticorpos, e a luz produzida pela excitação do fluoróforo fornece uma quantificação mais precisa de antígenos quando comparado com anticorpos secundários ligados à enzima. A sensibilidade e especificidade desta técnica podem ser aumentadas começando com proteínas imunoprecipitadas em vez de misturas brutas de proteína. Este procedimento sequencial é especialmente útil para a detecção de interações proteína-proteína. Por exemplo, a associação física de duas proteínas diferentes na membrana de um linfócito pode ser estabelecida pela imunoprecipitação de um extrato de membrana com o uso de um anticorpo específico para uma das proteínas e realizando um Western Blotting do imunoprecipitado usando um anticorpo específico para a segunda proteína que pode ter sido coimunoprecipitada juntamente com a primeira proteína. Uma variação da técnica de Western é usada rotineiramente para detectar a presença de anticorpos anti-HIV em soros de pacientes. Neste caso, uma mistura definida de proteínas HIV é separada por SDS-PAGE e transferida para uma membrana, e a membrana é incubada com diluições do soro de teste. A membrana é então analisada com uma segunda Ig anti-humana marcada para detectar a presença de anticorpos HIV-específicos que estavam no soro e se ligaram às proteínas HIV.
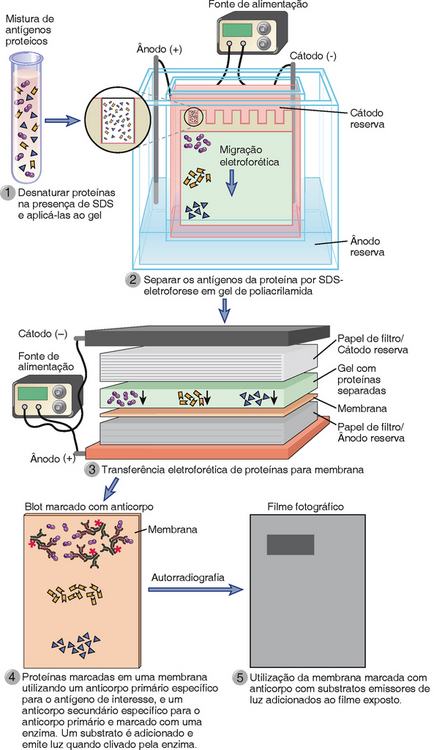
FIGURA A-3 Caracterização de antígenos por Western Blotting. Antígenos proteicos, separados por eletroforese de gel de poliacrilamida-dodecil sulfato de sódio (SDS) e transferidos para uma membrana, podem ser detectados por um anticorpo que por sua vez é revelado por um segundo anticorpo que pode ser conjugado a uma enzima como horsehadish peroxidase ou a um fluoróforo.
A técnica de transferir proteínas de um gel para uma membrana é chamada de Western Blotting como uma piada de bioquímicos. Southern é o sobrenome do cientista que primeiro transferiu DNA de um gel separador para uma membrana por transferência capilar, uma técnica chamada de Southern blotting desde então. Por analogia, Northern blotting foi o termo aplicado à técnica de transferir RNA de um gel para uma membrana, e Western Blotting é o termo usado para descrever a transferência de proteínas para uma membrana.
Marcação e Detecção de Antígenos em Células e Tecidos
Anticorpos específicos para antígenos expressos em ou dentro de tipos específicos de célula são comumente usados para identificar estas células em tecidos ou suspensões celulares e para separar estas células de populações mistas. Nestes métodos, o anticorpo pode ser radiomarcado, ligado à enzima, ou, mais comumente, marcados com fluorescência, e é usado um sistema de detecção que pode identificar o anticorpo ligado. Anticorpos ligados a contas magnéticas podem ser usados para isolar fisicamente células que expressam antígenos específicos.
Citometria de Fluxo e Separação por Fluorescência de Células Ativadas
A linhagem do tecido, estágio de maturação, ou status de ativação de uma célula pode ser determinado pela análise da superfície celular ou expressão intracelular de moléculas diferentes. Esta técnica é geralmente realizada pela coloração da célula com sondas marcadas por fluorescência que são específicas para aquelas moléculas e pela medição da quantidade de fluorescência emitida pela célula (Fig. A-4). O citômetro de fluxo é um instrumento especializado que pode detectar fluorescência em células individuais em uma suspensão e, assim, determinar o número de células expressando a molécula à qual uma sonda fluorescente se liga. Suspensões de células são incubadas com sondas marcadas por fluorescência, e a quantidade de sonda ligada por cada célula na população é medida pela passagem das células, uma de cada vez, através de um fluorímetro com um feixe incidente gerado por laser. As quantidades relativas de uma molécula específica em diferentes populações celulares podem ser comparadas pela coloração de cada população com a mesma sonda e pela determinação da quantidade de fluorescência emitida. Como preparação para análise por citometria de fluxo, suspensões celulares são coradas com as sondas fluorescentes de preferência. Mais frequentemente, estas sondas são anticorpos marcados com fluoróforos específicos para uma molécula na superfície celular. Alternativamente, moléculas citoplasmáticas podem ser coradas pela permeabilização temporária de células e pela permissão da entrada de anticorpos marcados através da membrana plasmática. Além dos anticorpos, diversos indicadores fluorescentes de concentrações de íon citoplasmático e de potencial de redução-oxidação podem ser detectados por citometria de fluxo. Estudos do ciclo celular podem ser realizados por análise de citometria de fluxo de células coradas com sondas fluorescentes ligantes de DNA como o iodeto de propídio. Células apoptóticas podem ser identificadas com sondas fluorescentes, como a anexina V, que se liga a fosfolipídios expostos anormalmente na superfície de células em processo de morte. Citômetros de fluxo modernos podem detectar três ou mais sinais fluorescentes de cores diferentes rotineiramente, cada um ligado a um anticorpo diferente ou a outra sonda. Esta técnica permite a análise simultânea da expressão de muitas combinações diferentes de moléculas por uma célula. Além da detecção de sinais fluorescentes, citômetros de fluxo também medem as propriedades de dispersão de luz frontal e lateral das células, que reflete o tamanho da célula e sua complexidade interna, respectivamente. Frequentemente, esta informação é usada para distinguir diferentes tipos de células. Por exemplo, comparado a linfócitos, neutrófilos causam maior dispersão lateral por causa de seus grânulos citoplasmáticos, e monócitos causam maior dispersão frontal devido a seu tamanho.
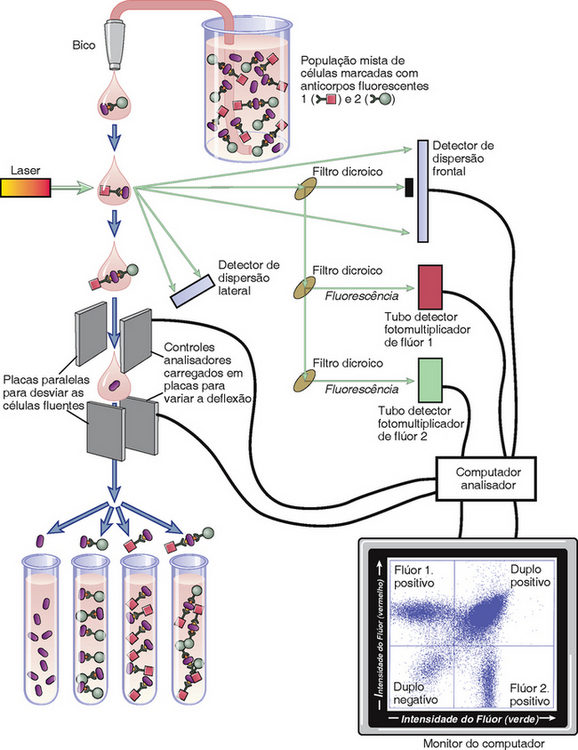
FIGURA A-4 Princípio da citometria de fluxo e separação de células ativada por fluorescência. O feixe de laser incidente tem comprimento de onda designado, e a luz que emerge da amostra é analisada quanto à dispersão frontal e lateral e quanto à luz fluorescente de dois ou mais comprimentos de onda que dependem dos marcadores fluoróforos ligados aos anticorpos. A separação descrita aqui é baseada em dois marcadores antigênicos (separação de duas cores). Instrumentos modernos podem analisar e separar populações celulares com base em sondas de três ou mais cores diferentes.
Purificação de Células
Um classificador por fluorescência de células ativadas é uma adaptação do citômetro de fluxo que permite a separação de células de uma população de acordo com quais e quantas sondas fluorescentes as células ligam. Esta técnica é efetuada pela deflexão diferencial das células com campos eletromagnéticos cuja força e direção são variadas de acordo com a intensidade medida do sinal fluorescente (Fig. A-4). As células podem ser marcadas com anticorpos rastreados com fluorescência ex vivo, ou, no caso de estudos experimentais com animais, a marcação pode ser efetuada in vivo pela expressão de transgenes que codificam proteínas fluorescentes, como a proteína verde fluorescente. (A tecnologia transgênica é descrita mais adiante neste apêndice.)
Outra técnica comumente usada para purificar células com um fenótipo específico depende de anticorpos que são ligados a contas magnéticas. Estes “reagentes imunomagnéticos” se ligam a certas células, dependendo da especificidade do anticorpo usado, e as células ligadas podem então ser puxadas para fora da suspensão por um ímã forte.
Imunofluorescência e Imuno-histoquímica
Anticorpos podem ser usados para identificar a distribuição anatômica de um antígeno dentro de um tecido ou dentro de compartimentos de uma célula. Para tanto, o tecido ou célula é incubado com um anticorpo marcado com um fluoróforo ou enzima, e a posição da marcação, determinada por um microscópio adequado, é usada para inferir a posição do antígeno. Na versão inicial deste método, chamado de imunofluorescência, o anticorpo era marcado com um corante fluorescente e podia se ligar a uma monocamada de células ou a uma seção congelada de um tecido. As células ou tecidos coloridos eram examinados com um microscópio de fluorescência para localizar o anticorpo. Embora sensível, o microscópio de fluorescência não é a ferramenta ideal para identificar as estruturas detalhadas de célula ou tecido devido a uma baixa razão de sinal/ruído. Este problema foi superado por novas tecnologias, incluindo a microscopia confocal, que usa tecnologia de seccionamento óptico para filtrar luz fluorescente desfocada, e a microscopia de dois fótons, que evita a formação de luz desfocada. Alternativamente, anticorpos podem ser ligados a enzimas que convertem substratos incolores em substâncias coloridas insolúveis que se precipitam na posição da enzima. Um microscópio de luz convencional pode ser usado para localizar o anticorpo em um célula ou tecido colorido. A variante mais comum deste método usa a enzima peroxidase horseradish, e o método é comumente chamado de técnica de imunoperoxidase. Outra enzima comumente usada é a fosfatase alcalina. Anticorpos diferentes ligados a enzimas diferentes podem ser usados em conjunto para produzir localizações simultâneas de duas cores de antígenos diferentes. Em outras variações, o anticorpo pode ser ligado a uma sonda com densidade de elétron como ouro coloidal, e a localização do anticorpo pode ser determinada em nível subcelular, uma técnica chamada de microscopia imunoeletrônica. Partícula de ouro com tamanhos diferentes foram usadas para a localização simultânea de antígenos diferente no nível ultraestrutural.
Em todos os métodos imunomicroscópicos, os sinais podem ser melhorados através do uso de técnicas do sanduíche. Por exemplo, em vez de ligar peroxidase horseradish a um anticorpo murino específico direcionado contra o antígeno de interesse, ele pode ser ligado a um segundo antianticorpo (p. ex., anticorpo Ig de coelho antimurino) que é usado para ligar o primeiro anticorpo, não marcado. Quando o marcador é ligado diretamente ao anticorpo primário, específico, o método é chamado de direto; quando o marcador é ligado a um anticorpo secundário ou terciário, o método é indireto. Em alguns casos, moléculas que não anticorpos podem ser usadas em métodos indiretos. Por exemplo, a proteína estafilocócica A, que se liga ao IgG, ou avidina, que se ligam ao anticorpos primários marcados com biotina, podem ser acoplados a fluoróforos ou enzimas.
Medição de Interações Antígeno-anticorpos
Em muitas situações, é importante conhecer a afinidade de um anticorpo por um antígeno. Por exemplo, a utilidade de um anticorpo monoclonal como reagente experimental ou terapêutico depende de sua afinidade. A afinidade de anticorpos por antígenos pode ser medida diretamente para antígenos pequenos (p. ex., haptenos) por um método chamado diálise de equilíbrio (Fig. A-5). Neste método, uma solução de anticorpo é confinada dentro de uma membrana “semipermeável” de celulose porosa imersa em uma solução contendo o antígeno. (Neste contexto, semipermeável significa que moléculas pequenas, como antígenos, conseguem passar livremente pelos poros da membrana, mas que macromoléculas, como anticorpos, não conseguem.) Se não há anticorpos presentes dentro do compartimento contido pela membrana, o antígeno na solução de banho entra até que a concentração do antígeno dentro do compartimento contido pela membrana seja exatamente a mesma que a de fora. Outra maneira de entender o sistema é que em equilíbrio dinâmico, antígenos entram e saem do compartimento contido pela membrana exatamente com a mesma taxa. Entretanto, quando há anticorpos presentes dentro da membrana, a quantidade total de antígenos dentro da membrana em ponto de equilíbrio aumenta pela quantidade que está ligada ao anticorpo. Este fenômeno ocorre porque apenas antígenos livres podem se difundir através da membrana, e no equilíbrio, é a concentração de antígenos livres que deve ser idêntica dentro e fora da membrana. A proporção do aumento de antígenos dentro da membrana depende da concentração de antígenos, da concentração de anticorpos e da constante de dissociação (Kd) da interação de ligação. Ao medir as concentrações de antígenos e anticorpos, pode-se calcular o Kd usando espectroscopia ou outros meios.
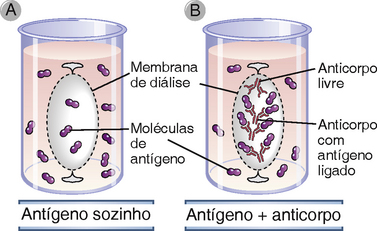
FIGURA A-5 Análise de ligação antígeno-anticorpo por diálise de equilíbrio. Na presença do anticorpo (B), a quantidade de antígeno dentro da membrana de diálise é aumentada comparada à ausência do anticorpo (A). Conforme descrito no texto, esta diferença, causado pela ligação de anticorpos de antígeno, pode ser usada para medir a afinidade do anticorpo pelo antígeno. Este experimento pode ser realizado somente quando o antígeno é uma molécula pequena (p. ex., um hapteno) capaz de cruzar a membrana de diálise livremente.
Uma maneira alternativa de determinar Kd é pela medição das taxas de formação e dissociação de complexos de antígeno-anticorpo. Estas taxas dependem, em parte, das concentrações de anticorpo e antígeno e da afinidade da interação. Todos os parâmetros exceto as concentrações podem ser resumidos como constantes, e tanto a constante on (Kon) como a constante off (Koff) podem ser calculadas experimentalmente pela determinação das concentrações e das taxas reais de associação ou dissociação, respectivamente. A proporção de Koff/Kon permite o cancelamento total de todos os parâmetros não relacionados à afinidade e é exatamente igual à constante de dissociação Kd. Assim, Kd pode ser medido por diálise de equilíbrio ou Kd pode ser calculado das constantes medidas sob condições de não equilíbrio.
Outro método, mais comumente usado atualmente, para medir a cinética de interações antígeno-anticorpo depende de ressonância de plasmon de superfície. Neste método, um instrumento biossensor especializado (como o Biacore) usa uma abordagem óptica para medir a afinidade de um anticorpo que é passado sobre um antígeno imobilizado sobre uma película metálica. Uma fonte de luz é focada nesta película por meio de um prisma a um ângulo específico (ressonância), e a luz refletida fornece uma leitura de ressonância de plasmon de superfície. A adsorção de um anticorpo para o antígeno altera a leitura de ressonância de plasmon de superfície, e esta alteração pode fornecer informações sobre a afinidade.
CAMUNDONGOS TRANSGÊNICOS E ALVOS GENÉTICOS
Três métodos importantes e relacionados para estudar os efeitos funcionais de produtos genéticos específicos in vivo são a criação de camundongos transgênicos convencionais que expressam um gene específico ectopicamente em um tecido definido; a criação de camundongos com genes “knockout”, nos quais uma interrupção alvo é usada para neutralizar a função de um gene específico; e a geração de camundongos “knockin”, nos quais um gene existente na linha germinativa é substituído por uma versão modificada do mesmo gene. Uma abordagem knockin poderia substituir uma versão normal do gene por uma versão mutante ou, em princípio, “corrigir” um gene mutante existente por uma versão “normal”. Estas técnicas, envolvendo camundongos geneticamente modificados, foram amplamente usadas para analisar muitos fenômenos biológicos, incluindo o desenvolvimento, ativação e tolerância de linfócitos.
Para a criação camundongos transgênicos convencionais, sequências estranhas de DNA, chamadas transgenes, são introduzidas nos pronúcleos de óvulos de camundongo fertilizados, e os óvulos são implantados nos ovidutos de fêmeas pseudográvidas. Geralmente, se algumas centenas de cópias de um gene são injetadas nos pronúcleos, aproximadamente 25% dos camundongos nascidos são transgênicos. De uma a 50 cópias do transgene se inserem em série em um local de quebra aleatória em um cromossomo e são subsequentemente herdados como um simples traço mendeliano. Como a integração normalmente ocorre antes da replicação do DNA, a maior parte (aproximadamente 75%) dos filhotes transgênicos carrega o transgene em todas suas células, inclusive nas células germinativas. Na maioria dos casos, a integração do DNA estranho não interrompe a função endógena do gene. Cada camundongo fundador portador do transgene é um heterozigoto, do qual linhas homozigóticas podem ser criadas.
O maior valor da tecnologia transgênica é que ela pode ser usada para expressar genes em tecidos específicos ao ligar sequências de codificação do gene às sequências reguladoras que normalmente direcionam seletivamente a expressão de genes naquele tecido. Por exemplo, promotores e intensificadores linfoides podem ser usados para super expressar genes, como os genes receptores de antígeno reorganizados, nos linfócitos, e o promotor de insulina pode ser usado para expressar genes nas células β das ilhotas pancreáticas. Exemplos da utilidade destes métodos no estudo do sistema imunológico são mencionados em diversos capítulos deste livro. Transgenes também podem ser expressos sob o controle de elementos promotores que respondem a fármacos ou hormônios, como a tetraciclina ou estrogênios. Nestes casos, a transcrição do transgene pode ser controlada à vontade pela administração do agente indutor.
Um método potente para o desenvolvimento de modelos animais de distúrbios de um único gene, e a maneira mais definitiva para estabelecer a função obrigatória de um gene in vivo, é a criação de camundongos knockout por mutação direcionada ou interrupção do gene. Esta técnica depende de fenômenos da recombinação homóloga. Por exemplo, se um gene exógeno é inserido em uma célula por eletroporação, ele pode se integrar aleatoriamente ao genoma da célula. No entanto, se o gene contém sequências que são homólogas a um gene endógeno, ele se combinará preferencialmente com e substituirá sequências endógenas. Para selecionar células que passaram pela recombinação homóloga, é usada uma estratégia de seleção baseada em fármacos. O fragmento do DNA homólogo a ser inserido em uma célula é colocado em um vetor que geralmente contem um gene resistente à neomicina e um gene (tk) de timidina cinase (Fig. A-6A). Esse vetor diretor é construído de tal forma que o gene resistente à neomicina está sempre inserido no DNA cromossômico, mas o gene tk gene é perdido sempre que a recombinação homóloga ocorre (ao contrário da inserção aleatória). O vetor é introduzido em células, e as células são desenvolvidas em neomicina e ganciclovir, um fármaco que é metabolizado pela timidina cinase para gerar um produto letal. Células nas quais o gene é integrado aleatoriamente serão resistentes à neomicina, mas serão mortas pelo ganciclovir, enquanto células nas quais ocorreu a recombinação homóloga serão resistentes aos dois fármacos porque o gene tk não será incorporado. Esta seleção positiva-negativa garante que o gene inserido em células sobrevivente passou pela recombinação homóloga com sequências endógenas. A presença do DNA inserido no meio de um gene endógeno normalmente interrompe as sequências codificadoras e neutraliza a expressão ou função daquele gene. Além disso, vetores direcionadores podem ser projetados de modo que a recombinação homóloga leve à deleção de um ou mais éxons do gene endógeno.
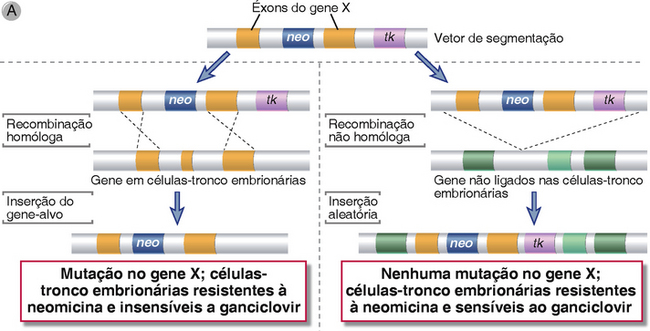
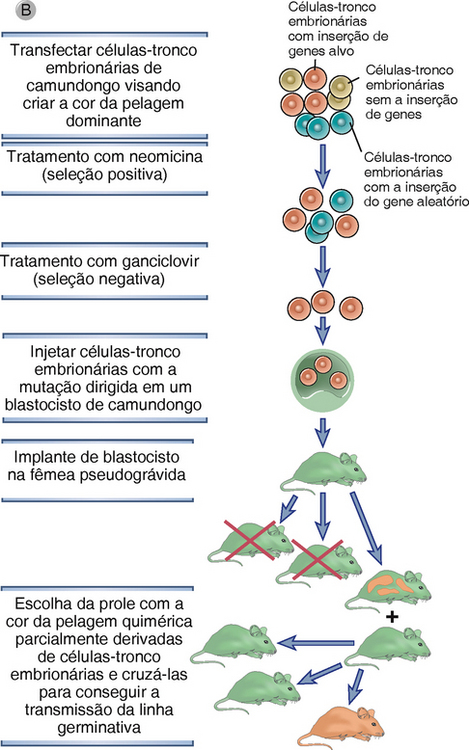
FIGURA A-6 Geração de knockout de gene. A, A interrupção do gene X em uma célula estaminal embrionária (ES) é realizada por recombinação homóloga. Uma população de células ES é transfectada com um vetor direcionador que contém sequências homólogas aos dois éxons do gene X acompanhando um gene de resistência à neomicina (neo). O gene neo substitui ou interrompe um dos éxons do gene X na recombinação homóloga. O gene timidina cinase (tk) no vetor será inserido no genoma somente se ocorrer uma não recombinação homóloga, aleatória. B, As células ES que foram transfectadas pelo vetor direcionador são selecionadas por neomicina e ganciclovir de modo que apenas as células com inserção alvejada (recombinação homóloga) sobrevivem. Estas células são então injetadas em um blastocisto, que é depois implantado no útero de um camundongo pseudográvido. Um camundongo quimérico se desenvolverá, no qual alguns dos tecidos são derivados da célula ES portadora da mutação alvejada no gene X. Estes camundongos quiméricos são identificados pela pelagem de cor mista, incluindo a cor da linhagem de camundongo da qual as células ES foram derivadas e a cor da linhagem de camundongo da qual o blastocisto foi derivado. Se a mutação estiver presente em células germinais, ela pode ser propagada por mais procriações.
Para gerar um camundongo portador de uma interrupção ou mutação do gene direcionado, um vetor direcionador é usado para primeiro interromper o gene em uma linhagem celular estaminal embrionária (ES) murina. Células ES são células pluripotentes derivadas de embriões de camundongo que podem ser propagadas e induzidas a se diferenciar em uma cultura ou que podem ser incorporadas em um blastocisto de camundongo, que pode ser implantado em uma mãe pseudográvida e levado a termo. O mais importante é que a progênie das células ES se desenvolve normalmente em tecidos maduros que vão expressar os genes exógenos que foram transfectados para as células ES. Assim, o vetor direcionador projetado para interromper um gene específico é inserido em células ES, e colônias onde ocorreu a recombinação homóloga (em um cromossomo) são selecionadas com fármacos, conforme descrito anteriormente (Fig. A-6B). A presença da recombinação desejada é verificada pela análise do DNA com técnicas como a hibridização com Southern blot ou reação em cadeia de polimerase. As células ES selecionadas são injetados em blastocistos, que são implantados em fêmeas pseudográvidas. Camundongos que se desenvolvem serão quiméricos para uma interrupção ou mutação heterozigótica, isto é, alguns dos tecidos serão derivados das células ES e outros do resto do blastocisto normal. As células germinativas geralmente também são quiméricas, mas como estas células são haploides, apenas algumas conterão a cópia do cromossomo com o gene interrompido (mutado). Se camundongos quiméricos forem cruzados com animais normais (tipo selvagem) e esperma ou óvulos contendo o cromossomo com a mutação se fundir com o parceiro do tipo selvagem, todas as células na progênie derivadas de tal zigoto serão heterozigóticas para a mutação (chamada de transmissão de linhagem germinal). Tais camundongos heterozigóticos podem ser cruzados para produzir animas que serão homozigóticos para a mutação com uma frequência previsível por segregação mendeliana simples. Estes camundongos knockout são deficientes na expressão do gene-alvo.
A recombinação homóloga também pode ser usada para substituir uma sequência normal de genes por uma versão modificada do mesmo gene (ou de outro gene), criando dessa maneira uma linhagem de camundongos knockin. Camundongos knockin podem ser usados para avaliar as consequências biológicas de uma alteração em apenas uma base, por exemplo, ao contrário da deleção de um gene. Em princípio, uma abordagem knockin também poderia ser usada para substituir um gene defeituoso por um normal. Em certas circunstâncias, um gene diferente pode ser posicionado em um local definido no genoma pelo uso de uma estratégia de knockin em vez de ser posicionado aleatoriamente como ocorre em camundongos transgênicos convencionais. Abordagens knockin são usadas quando é desejável ter a expressão do transgene regulada por certas sequências endógenas de DNA, como um intensificador específico ou uma região promotora. Neste caso, o vetor direcionador contém um gene exógeno codificando um produto desejado e sequências homólogas para um gene endógeno que são necessárias para alvejar o local da recombinação.
Embora a estratégia tradicional de alvo genético tenha sido de grande utilidade em pesquisas imunológicas, esta abordagem tem algumas limitações. Primeiro, a mutação de um gene durante o desenvolvimento pode ser compensada pela expressão alterada de outros produtos genéticos, e, portanto a função do gene alvo pode ser obscurecida. Segundo, em um camundongo com gene knockout convencional, a importância de um gene em apenas um tecido ou em um único momento durante o desenvolvimento não pode ser facilmente avaliada. Terceiro, um gene marcado seletivo funcional, como o gene de resistência à neomicina, é permanentemente introduzido no genoma do animal, e esta alteração pode ter resultados imprevisíveis no fenótipo do animal. Um aperfeiçoamento importante da tecnologia de knockout de gene que pode superar muitas destas desvantagens é uma abordagem de alvo “condicional”. Uma estratégia condicional comumente usada tira proveito do sistema de recombinação derivado de bacteriófago Cre/loxP. A enzima Cre é uma recombinase de DNA que reconhece um padrão de sequência 34-bp chamado loxP, e a enzima media a deleção de segmentos de gene acompanhados por dois locais de loxP na mesma orientação. Para gerar camundongos com genes marcados com loxP, vetores direcionadores são construídos com um local loxP acompanhando o gene de resistência à neomicina em uma extremidade e um segundo local loxP acompanhando as sequências homólogas ao alvo na outra extremidade. Esses vetores são transfectados para células ES, e camundongos portadores do gene alvo acompanhado por loxP, mas ainda funcional são gerados do modo descrito para camundongos knockout convencionais. Uma segunda linhagem de camundongos portadores de um transgene cre é então criada com a linha portadora do gene alvo acompanhado por loxP. Na progênie, a expressão de Cre recombinase mediará a deleção do gene alvo. Tanto as sequências normais de genes quanto a resistência à neomicina serão eliminadas. O mais importante é que a expressão do gene cre, e, portanto a deleção do gene alvo, pode ser restrita a certos tecidos ou momentos especificados pelo uso de construtos transgene cre com promotores diferentes. Por exemplo, a deleção seletiva de um gene somente em células T auxiliares (helper) pode ser alcançada pelo uso de um camundongo cre transgênico no qual cre é estimulada por um promotor CD4. Alternativamente, um promotor induzível por esteroide pode ser usado para que a expressão de Cre e a deleção subsequente do gene ocorram somente depois dos camundongos receberem uma dose de dexametasona. Muitas outras variações foram criadas para esta tecnologia, para criar mutantes condicionais. A tecnologia Cre/loxP também pode ser usada para criar camundongos knockin. Neste caso, locais loxP são colocados no vetor direcionador para acompanhar o gene de resistência à neomicina e as sequências homólogas, mas eles não acompanham as sequências de substituição de gene (knockin). Assim, depois da deleção mediada por cre, o gene exógeno permanece no genoma no local alvo.
MÉTODOS PARA O ESTUDO DE RESPOSTAS DE LINFÓCITOS T
Nosso conhecimento atual sobre os eventos celulares na ativação de células T é baseado em uma variedade de técnicas experimentais nas quais diferentes populações de células T são ativadas por estímulos definidos e respostas funcionais são mensuradas. Experimentos in vitro forneceram uma grande quantidade de informações sobre as mudanças que ocorrem em uma célula T quando ela estimulado por antígeno. Mais recentemente, foram desenvolvidas diversas técnicas para estudar a proliferação de células T, a expressão de citocinas, e a redistribuição anatômica em respostas à ativação do antígeno in vivo. As novas abordagens experimentais têm sido especialmente úteis para o estudo da ativação de células T virgens (naïve) e para a localização de células T de memória antígeno-específicas após a atenuação de uma resposta imunológica.
Ativação Policlonal de Células T
Ativadores policlonais de células T se ligam a muitos ou a todos os complexos receptores de células T (TCR) independentemente da especificidade e ativam as células T de maneiras semelhante aos complexos peptídeo-MHC em células apresentadoras de antígenos (APC). Os ativadores policlonais são usados majoritariamente in vitro para ativar células T isoladas de sangue humano ou de tecidos linfoides de animais experimentais. Ativadores policlonais também podem ser usados para ativar células T com especifidades a antígeno desconhecidas, e podem evocar uma resposta detectável de populações mistas de células T virgens, mesmo que a frequência de células específicas para qualquer antígeno fosse baixa demais para despertar uma resposta detectável. As proteínas vegetais poliméricas que se ligam a carboidratos, chamadas lectinas, como a concanavalina-A e a fito-hemaglutinina, são um grupo de ativadores de células T comumente usado. Estas lectinas se ligam especificamente a certos resíduos de açúcar nas glicoproteínas da superfície de células T, incluindo as proteínas TCR e CD3, estimulando assim as células T. Anticorpos específicos para epitopos de estrutura invariável em proteínas TCR e CD3 também funcionam com ativadores policlonais de células T. Com frequência, estes anticorpos precisam ser imobilizados em superfícies sólidas ou contas ou formar ligações cruzadas com antianticorpos secundários para induzir respostas de ativação ótimas. Como os ativadores policlonais solúveis não fornecem sinais coestimuladores que são normalmente fornecidos pelas APC, eles frequentemente são usados junto com anticorpos estimuladores para receptores de coestimuladores, como antiCD28 ou antiCD2. Superantígenos, outro tipo de estímulo policlonal, se ligam a e ativam todas as células T que expressam tipos específicos de cadeia β de TCR (Cap. 15, Fig. 15-2). Células T de qualquer especificidade de antígeno também podem ser estimuladas com reagentes farmacológicos, como a combinação do éster de forbol PMA e o ionóforo de cálcio ionomicina, que imitam os sinais gerados pelo complexo TCR.
Ativação Induzida por Antígeno de Populações de Células T Policlonais
Populações policlonais de células T normais que são enriquecidas para células T específicas para um antígeno específico podem ser derivadas do sangue e de órgãos linfoides periféricos de indivíduos depois de imunização com o antígeno. A imunização serve para expandir o número de células T antígeno-específica, que podem então ser reestimuladas in vitro pela adição de antígeno e APC combinadas a MHC para as células T. Esta abordagem pode ser usada para estudar a ativação induzida de antígeno de uma população mista de células T ativadas anteriormente (“primed”) expressando muitas TCRs diferentes, mas o método não permite a análise de respostas de células T virgens.
Ativação Induzida por Antígeno de Populações de Células T com Especificidade de um Único Antígeno
Populações monoclonais de células T, que expressam TCR idênticos, têm sido úteis nas análises funcionais, bioquímicas e moleculares. A limitação destas populações monoclonais é que elas são mantidas como linhagens de cultura de tecido de longo termo e, portanto podem divergir fenotipicamente de células T normais in vivo. Um tipo de população de células T monoclonais que é usado frequentemente em imunologia experimental é um clone de células T antígeno-específicas. Tais clones são derivados pelo isolamento de células T de indivíduos imunizados, como descrito para células T policlonais, seguido por estimulação in vitro repetitiva com o antígeno imunizador mais APC combinadas com MHC e a clonagem de células antígeno–responsivas únicas em meios semissólido ou em meios líquidos por diluição limitadora. Respostas antígeno-específicos podem ser facilmente mensuradas nestas populações porque todas as células em uma linhagem de células clonadas têm os mesmos receptores e foram selecionadas para crescimento em resposta a um complexo antígeno-MHC conhecido. Clones de linfócitos T auxiliares e citotóxicos foram estabelecidos de camundongos e de humanos. Outras populações de células T monoclonais usadas no estudo da ativação de células T incluem hibridomas de células T antígeno-específicos, que são produzidos como hibridomas de células B (Fig. 5-9, Cap. 5), e linhagens de tumor derivadas de células T foram estabelecidas in vitro após a remoção de células T malignas de animais ou humanos com leucemias ou linfomas em células T. Apesar de algumas linhagens derivadas de tumor expressarem complexos TCR funcionais, suas especificidades de antígeno não são conhecidas, e as células são geralmente estimuladas com ativadores policlonais para fins experimentais. A linhagem Jurkat, derivada de uma célula de leucemia humana de célula T, é um exemplo de uma linhagem de tumor amplamente usada como modelo para o estudo da transdução de sinal de células T.
Camundongos transgênicos TCR são uma fonte de células T homogêneas, fenotipicamente normais com especificidades de antígeno idênticas que são amplamente usadas para análises experimentais in vitro e in vivo. Se os genes da cadeia α e β de uma única TCR de especificidade conhecida são expressos como um transgene em camundongos, uma maioria das células T maduras nos camundongos expressará aquela TCR. Se o transgene TCR é cruzado em um background deficiente em RAG-1 ou RAG-2, não ocorre nenhuma expressão de gene TCR endógeno e 100% das células T expressarão somente o TCR transgênico. Células T transgênicas TCR podem ser ativadas in vitro ou in vivo com um único antígeno peptídeo, e elas podem ser identificadas por anticorpos específicos para o TCR transgênicos. Uma das vantagens de camundongos transgênicos TCR é que eles permitem o isolamento de números suficientes de células T virgens de especificidade definida que possibilitam o estudo de respostas funcionais à primeira exposição ao antígeno. Esta vantagem permitiu que investigadores estudassem as condições in vitro sob as quais a ativação de antígeno de células T virgens leva à diferenciação em subtipos funcionais como células TH1 e TH2 (Cap. 9). Células T virgens de camundongos transgênicos TCR também podem ser injetadas em camundongos receptores singenêicos normais, onde elas migram para tecidos linfoides. O camundongo receptor é então exposto ao antígeno para o qual a TCR transgênica é específica. O uso de anticorpos que marcam as células T TCR transgênicas permite que sua expansão e diferenciação in vivo sejam seguidas, e que possam ser isoladas para análise de respostas de memória (secundárias) ao antígeno ex vivo.
Métodos para Enumerar e Estudar Respostas Funcionais de Células T
Ensaios de proliferação para linfócitos T, como aqueles de outras células, são conduzidos in vitro pela determinação da quantidade de timidina 3H-marcada incorporada no DNA reprodutor de células cultivadas. A incorporação de timidina fornece uma medida quantitativa da taxa de síntese de DNA, que geralmente é diretamente proporcional à taxa de divisão celular. A proliferação celular in vivo pode ser medida pela injeção o análogo de timidina, bromodeoxiuridina (BrdU) em animais e corando as células com anticorpo anti-BrdU para identificar e enumerar núcleos que incorporaram BrdU em seu DNA durante a reprodução de DNA.
Corantes fluorescentes podem ser usados para estudar a proliferação de células T in vivo. As células T são primeiro marcadas com ésteres fluorescentes lipofílicos quimicamente reativos e depois transferidas para animais experimentais. Os corantes entram nas células, formam ligações covalentes com proteínas citoplasmáticas, e depois não conseguem sair das células. Um corante deste tipo usado comumente é o 5,6-diacetato carboxifluoresceína succinimidil éster (CFSE), que pode ser detectado em células por técnicas padrão de citometria de fluxo. Toda vez que uma célula T se divide, seu conteúdo de corante é dividido, e é possível determinar se as células T transferidas presentes em tecidos linfoides do camundongo receptor se dividiram in vivo e estimar o número de duplicações pelas quais cada célula T passou.
Tetrâmeros peptídeo-MHC são usados para enumerar células T com uma única especificidade antígeno isolada do sangue ou de tecidos linfoides de animais experimentais ou de humanos. Estes tetrâmeros contêm quatro dos complexos peptídeo-MHC que as células T normalmente reconheceriam na superfície das APCs. O tetrâmero é feito pela produção de uma molécula MHC classe I à qual uma pequena molécula chamada biotina é ligada pelo uso de tecnologia do DNA recombinante. A biotina se liga com alta afinidade a uma proteína chamada avidina, e cada molécula de avidina se liga a quatro moléculas de biotina. Assim, a avidina forma um substrato para reunir quatro proteínas MHC conjugadas à biotina. As moléculas MHC podem ser carregadas com um peptídeo de interesse e estabilizadas desta maneira, e a molécula de avidina é marcada com um fluoróforo, como FITC. Este tetrâmero se liga a células T específicas para o complexo peptídeo-MHC com avidez alta o suficiente para marcar as células T mesmo em suspensão. Este método é a única abordagem viável para a identificação de células T antígeno-específicas em humanos. Por exemplo, é possível identificar e numerar células T circulantes HLA-A2 restritas específicas para um peptídeo HIV corando células sanguíneas com um tetrâmero de moléculas HLA-A2 carregado com o peptídeo. A mesma técnica está sendo usada para enumerar e isolar células T específicas para autoantígenos em indivíduos normais e em pacientes com doenças autoimunes. Tetrâmeros peptídeo-MHC que se ligam a um TCR transgênico específico podem ser usados para quantificar as células T transgênicas em diferentes tecidos após transferência adotiva e estimulação de antígeno. Atualmente a técnica é amplamente usada com moléculas MHC de classe I; em moléculas classe I, apenas um polipeptídio é polimórfico, e moléculas estáveis podem ser produzidas in vitro. Isso é mais difícil para moléculas classe II porque as duas cadeias são polimórficas e necessárias para a montagem adequada, mas tetrâmeros de peptídeos classe II também estão sendo produzidos.
Ensaios de secreção de citocina podem ser usados para quantificar células T efetoras secretoras de citocina dentro de tecidos linfoides. Os métodos mais comumente usados são a coloração citoplasmática de citocinas de ensaios imunoabsorventes de célula única ligada à enzima (ELISpot). Nestes tipos de estudos, a ativação e diferenciação de células T induzida por antígenos ocorre in vivo, e então as células T são isoladas e testadas para a expressão de citocina in vitro. A coloração citoplasmática de citocinas exige a permeabilização de células para que os anticorpos específicos marcados com fluoróforo possam entrar na célula, e as células coradas analisada por citometria de fluxo. A expressão de citocinas por células T específicas para um antígeno específico pode ser determinada por coloração adicional de células T com tetrâmeros peptídeo-MHC ou, no caso de células T transgênicas TCR, anticorpos específicos para o TCR transgênico. Usando uma combinação de CFSE e anticorpos anticitocina, é possível examinar a relação entre a divisão celular e a expressão de citocinas. No ensaio ELISpot, células T recém isoladas do sangue ou de tecidos linfoides são cultivadas em poços plásticos revestidos com anticorpos específicos para uma citocina específica. Conforme as citocinas são secretadas de células T individuais, elas se ligam aos anticorpos em pontos discretos correspondentes à localização de células T individuais. Os pontos são visualizados pela adição de anti-Ig ligado à enzima, como em um ELISA padrão (veja anteriormente), e o número de pontos é contado para determinar o número de células T secretoras de citocina.
MÉTODOS PARA O ESTUDO DE RESPOSTAS DE LINFÓCITOS B
Ativação de Populações de Células B Policlonais
É tecnicamente difícil estudar os efeitos de antígenos sobre células B normais porque, como previsto pela hipótese de seleção clonal, pouquíssimos linfócitos em um indivíduo são específicos para qualquer antígeno. Uma abordagem para sair desse problema é usar anticorpos anti-Ig como análogos de antígenos, com a suposição que o anti-Ig se ligará a regiões constantes (C) de moléculas Ig de membrana em todas as células B e terão os mesmos efeitos biológicos como o antígeno que se liga a regiões hipervariáveis de moléculas Ig de membrana apenas nas células B antígeno-específicas. Até o ponto em que comparações exatas são viáveis, essa suposição parece geralmente correta, indicando que o anticorpo anti-Ig é um modelo válido para antígenos. Consequentemente, o anticorpo anti-Ig é frequentemente usado como ativador policlonal de linfócitos B, semelhante ao uso de anticorpos anti-CD3 como ativadores policlonais de linfócitos T discutidos anteriormente.
Ativação Induzida por Antígeno de Populações de Células B com Especificidade de um Único Antígeno
Para examinar os efeitos da ligação de antígenos a células B, investigadores tentaram isolar células B antígeno-específicas de populações complexas de linfócitos normais ou para produzir células clonadas de linhagens B com especificidades antigênicas definidas. Estes esforços tiveram pouco sucesso. No entanto, foram desenvolvidos camundongos transgênicos nos quais quase todas as células B expressam um Ig transgênico de especificidade conhecida, de modo que a maior parte das células B nestes camundongos responde ao mesmo antígeno. Uma abordagem um pouco mais sofisticada foi a geração de camundongos knockin para receptores de antígeno, nos quais genes de cadeia Ig H e L reorganizados tiveram recombinação homóloga em seus loci endógenos. Tais animais knockin demonstraram ser particularmente úteis na análise de edição do receptor.
Ensaios para Medir Proliferação de Célula B e Produção de Anticorpos
Grande parte de nosso conhecimento sobre a ativação de células B é baseado em experimento in vitro, onde estímulos diferentes são usados para ativar células B, e sua proliferação e diferenciação podem ser mensuradas precisamente. Os mesmos ensaios podem ser feitos com células B recuperadas de camundongos expostos a antígenos diferentes com células B homogêneas que expressam receptores de antígeno codificados para transgenes.
A proliferação de células B é mensurada pelo uso de marcação CFSE ou incorporação de timidina 3H-marcada in vitro e marcação com BrdU in vivo, conforme já descrito para a proliferação de células T.
A produção de anticorpos é mensurada de duas maneiras diferentes: com ensaios para secreção cumulativa de Ig, que medem a quantidade de Ig que se acumula no sobrenadante de linfócitos cultivados ou no soro de um indivíduo imunizado; e com ensaios de célula única, que determinam o número de células em uma população imune que secreta Ig de uma especificidade ou isótipo específico. A técnica mais precisa, quantitativa e amplamente usada para medir a quantidade total de Ig em uma cultura sobrenadante ou amostra de soro é a ELISA. Através do uso de antígenos ligados a suportes sólidos, é possível usar ELISA para quantificar a quantidade de anticorpo em uma amostra específica para um antígeno específico. Além disso, a disponibilidade de anticorpos anti-Ig que detectam Igs de categorias diferentes, pesadas ou leves, permite a medição das quantidades de isótopos diferentes em uma amostra. Outras técnicas para medir os níveis de anticorpo incluem a hemaglutinação para anticorpos antieritrócitos e lise dependente de complemento para anticorpos específicos para tipos de célula conhecidos. Ambos os ensaios são baseados na demonstração de que se a quantidade de antígeno (i. e., células) é constante, a concentração do anticorpo determina a quantidade de anticorpo ligado às células, e isso é refletido no grau de aglutinação das células ou na ligação subsequente de lise de complemento e de célula. Resultados destes ensaios são normalmente expressos como títulos de anticorpos, que são a diluição da amostra dando efeitos na metade do máximo ou a diluição na qual o ponto final do ensaio é alcançada.
Um ensaio de célula única para secreção de anticorpos é a análise ELISpot. Neste método, o antígeno é ligado ao fundo de um poço, células secretoras de anticorpos são adicionadas, e os anticorpos que foram secretados e estão ligados ao antígeno são detectados por um anticorpo anti-Ig ligado à enzima, como na análise ELISA, em um meio semissólido. Cada ponto representa a localização de uma célula secretora de anticorpos. Ensaios de célula única fornecem uma medida dos números de células secretoras de Ig, mas não podem quantificar precisamente a quantidade de Ig secretada por cada célula ou pela população total. As técnicas ELISA e ELISpot podem ser adaptadas para avaliar a afinidade de anticorpos, pelo uso de antígenos com números diferentes de grupos haptênicos. Desta maneira, a maturação da afinidade pode ser avaliada pela testagem do soro ou de células B amostradas em momentos diferentes durante uma resposta imunológica.